Dans les quatre parties précédentes de cette série estivale, nous avons étudié les différentes techniques de séquençage de l’ADN humain (ou pas).
Nous allons maintenant passer à la partie numérique de la question en faisant le tour des outils informatiques qui exploitent les données brutes du séquençage pour reconstituer l’ADN, et les applications qui en résultent car c’est là l’essentiel. La discipline de la bio-informatique qui se développe à très grande vitesse est immense et ne sera que très partiellement couverte dans ces articles. On verra comment des acteurs tels que Google ou nVidia sont impliqués dans ces sujets.
Un peu de vocabulaire avant la route du numérique
Pour mémoire, le séquençage complet du génome humain a été réalisé en 2003 par le consortium international “Human Genome Project”. Le projet avait démarré en 1989 et était financé par le Department of Healthcare US et aussi par le Department of Energy. Ce dernier est intéressé par la question pour ses applications dans la production d’énergies vertes comme les biofuels. Le projet HGP devait durer 15 ans mais un séquençage brut complet à lus de 90% avait été terminé fin 2000 et publié début 2001. C’est seulement en 2004 qu’a été publié un séquençage complet du génome humain qui s’appuie sur la compilation de séquences d’ADN de plusieurs personnes distinctes (en bonne santé). L’histoire et l’étendue de la dimension technique du Human Genome Project est très bien expliquée dans ce long papier de 62 pages publié en 2001 dans la revue Nature.
C’est seulement en 2007 que les séquenceurs de seconde génération ont permis à un cout plus raisonnable de réaliser le séquençage d’une seule personne. Cela a permis de déterminer que la différence entre deux ADN humains était d’environ une base pour mille. On appelle cela les “SNP” ou Single Nucleotide Polymorphisms, les variations de l’ADN par rapport à une référence qui est celle du Human Genome Project. Ce taux de variation est différent selon les espèces vivantes.
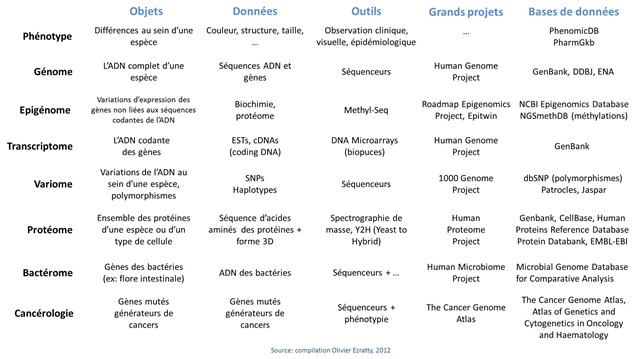
Ce qui m’amène à évoquer quelques termes et domaines de recherche de génétique et de biochimie qui seront cités par la suite :
- Phenotype : caractérise les variations au niveau des membres d’une même espèce. Dans l’espèce humaine, cela correspond à la couleur de la peau, à la taille, à la forme, à la couleur des yeux et aux maladies. Chaque personne a en moyenne 200 à 300 déficiences dans son génome et qui sont observables dans son phénotype !
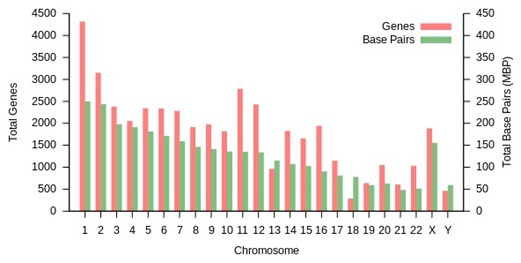
- Génotype : c’est l’ADN et les gènes qu’elle contient. Pour mémoire, 3 milliards de paires de bases dans l’ADN humain (certains disent 6 milliards, qui correspondent à l’ADN après la duplication des brins qui précède la division cellulaire, on parle alors d’ADN diploïde par opposition à l’ADN haploïde, non répliquée). A noter que l’on ne connait pas encore tous les gènes de l’espèce humaine. Le nombre de gènes a pas mal varié dans le temps pour monter jusqu’à 100000 avant le Human Genome Project. Il est maintenant estimé aux alentours de 32000 sachant qu’un peu plus de 20000 gènes ont été identifiés et décodés à ce jour. Il en reste 10000 à trouver dans le flot d’ADN de nos 3 milliards de bases ! Ci-dessous, la répartition des bases et gènes par chromosome (source : Wikipedia).
- Polymorphisme : ce sont les légères variations dans l’ADN des individus, de l’ordre de 1 pour 1000 bases, soit environ 3 millions de bases, et qui sont détectées lors du séquençage complet d’un génome humain ou, le plus souvent, avec des techniques à base de marqueurs et bio-puces (DNA Array). La référence utilisée est celle du Human Genome Project. Un nouveau vaste projet a été lancé en 2008 : le 1000 Genome Project pour identifier les polymorphismes que l’on trouve dans au moins 1% de la population des 2500 individus qui fait l’objet de l’étude. La population étudiée couvre toute la diversité de l’espèce humaine (tous continents, tous âges, et les deux sexes). Les données générées par ce projet sont publiques et exploitables par tous les laboratoires de recherche.
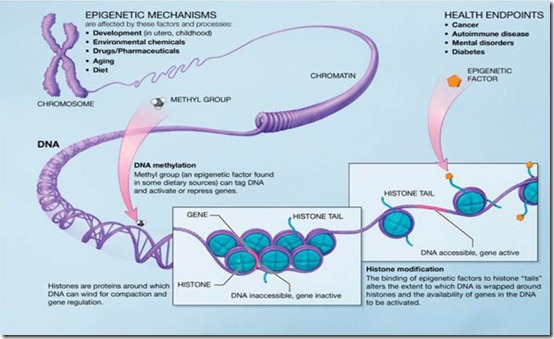
- Epigénome : facteurs externes à l’ADN qui expliquent les variations dans l’expression des gènes au sein d’une espèce vivante. Ces facteurs sont d’origine chimique : l’alimentation, les médicaments, la pollution. L’épigénome est en quelque sorte la caractérisation de l’environnement sur le fonctionnement chimique des cellules. Au niveau de la molécule d’ADN, les marqueurs épigénétiques principaux relèvent de la méthylation de l’ADN, une transformation chimique des bases qui va affecter la manière dont les parties codantes de l’ADN vont pouvoir s’exprimer, notamment avec la mécanique cellulaire de fabrication des protéines comme les ARN de transfert ou les ribosomes. Il y a aussi les modifications qui peuvent intervenir au niveau des histones, ces molécules autour desquelles les doubles brins d’ADN s’enroulent. Ces modifications vont affecter la manière dont l’ADN va s’enrouler autour des histones et par là, modifier l’expression des gènes qui correspond à la partie de l’ADN qui n’est pas enroulée autour d’histones (revoir le premier article pour mieux comprendre). L’épigénétique est la science d’étude de l’épigénome. On peut évaluer les parties de l’ADN qui sont sur ou sous-méthylées avec des systèmes tels que Methyl-Seq d’Agilent qui traitent chimiquement l’ADN avant son séquençage. Cela sert notamment aux recherches sur la propagation des cancers. On peut aussi étudier la manière dont les chromosomes sont repliés sur eux-mêmes et les liaisons qui peuvent se créer entre plusieurs morceaux d’ADN. C’est l’objet de la méthode de préparation de séquençage par ligase appelée Hi-C Seq. Elle sert à identifier les liens entre les zones des chromosomes.
- Transcriptome : c’est la partie de l’ADN qui est transcrite en séquences codantes (cDNA) puis dans les différents ARN. Le transcriptome caractérise une partie de l’expression des gènes sachant que celle-ci dépend aussi de facteur épigénétiques que nous venons de voir.
- Protéome : protéines générées par les cellules, qui varient à la fois en fonction de l’expression des gènes (transcriptome) mais aussi de phénomènes externe (épigénome). Un même gène donné peut coder plusieurs variantes d’une même protéine du fait de modifications des ARN messagers lors de leur processus de maturation avant la création des protéines via les ribosomes, mais aussi à des modifications des protéines suite à leur production (phosphorylations, glycosylations).
- Bactérome : décrit l’écosystème de bactéries d’une espèce vivante. Pour l’homme, il s’agit des bactéries internes, dans dans la flore intestinale (l’essentiel), les cavités bucales et nasales, les organes génitaux ainsi qu’externes, sur la peau. A noter le Human Microbiome Project, financé à hauteur de $150 sur 5 ans (2009-2014) par l’équivalent américain de l’INSERM (le National Institute of Health, ou NIH), qui vise à séquencer le bactérome humain complet en s’appuyant sur des échantillons prélevés sur 250 personnes différentes. Cela représente plus de 10000 bactéries différentes et plus d’une centaine de fois le nombre de bases du génome humain. C’est un autre projet gigantesque qui vise à identifier la corrélation entre le bactérome et les différentes pathologies qui nous affectent et notamment l’obésité ou le psoriasis.
Les étapes numériques du séquençage
Le cout “physique” du séquençage du génome humain, et de l’ADN et de l’ARN en général, a baissé plus rapidement que ne le permet la loi de Moore ces dernières années. Ainsi, il était de $500m il y a une douzaine d’année, de quelques centaines de milliers de dollars au milieu des années 2000 et il est aujourd’hui inférieur à $1000 et s’apprête à descendre en dessous de $100. Je vous recommande au passage la lecture du document $100 genome implications for the DoD (décembre 2010) qui couvre cette question ainsi que les implications pratiques de la baisse du coût du séquençage du génome humain.
$100, oui. Mais à un détail près : le traitement numérique des données du séquençage ! Et celui-ci est en train de devenir supérieur à celui du séquençage proprement dit car il est extrêmement complexe. Sa complexité va dépendre de la connaissance préalable que l’on de l’ADN séquencé : s’il s’agit d’un ADN humain, on pourra s’appuyer sur les données du séquençage réalisé dans le cadre du Human Genome Project, ce qui sera très utile pour simplifier les calculs. S’il s’agit par contre d’un ADN complètement nouveau comme celui d’une espèce vivante ou animale ou d’une bactérie encore non étudiée, on partira alors de zéro (séquençage “di novo”) et la tâche sera plus lourde.
Parcourons maintenant les différentes étapes de ce traitement numérique :
- Génération des séquences d’ADN dans les séquenceurs
Les séquenceurs génèrent d’abord une donnée brute qui est souvent une suite d’images liées aux capteurs optiques (SOLID, Illumina, Roche 454, Pacific Bioscience) ou non optiques (Ion Torrent, GeniaChip, Stratos Genomics, Oxford Nanopore). Elles correspondent à une base d’ADN détectée dans plusieurs zones (microcuves, nanopores, plaque, etc) qui sont liées chacune à un brin d’ADN.
Ces images sont généralement stockées au format TIFF ce qui représenterait 30 To de données pour un génome humain. C’est dans le PC du séquenceur qu’est effectuée la conversion à la volée de ces images en séquences d’ADN (“base call”), ce qui génère 100 Go de données, ce qui est déjà plus abordable et permet d’éviter de stocker 30 To ! Pourquoi 100 Go ? A cause du taux de couverture du séquençage qui voit l’ADN explosé (shotgun) en petits morceaux de taille et positionnement aléatoires – de 100 à 1000 bases en général – et avec une redondance d’un facteur qui va jusqu’à 40. 100 Go revient à un taux de couverture de x33. Ce taux peut baisser quand la taille moyenne des brins séquencés augmente. Ce qui réduira d’ailleurs aussi la charge de calcul par la suite. Les 100 Go comprennent aussi des données sur la qualité du séquençage (probabilité d’erreur pour chaque base). Le format de stockage exploite 1 octet par base (qui tient en théorie sur 2 bits et non 8 puisqu’il n’y a que quatre bases différentes). Il dépend des constructeurs. Chez Illumina, il s’agit d’un format binaire d’extension .bcl.
- Alignement des séquences
Ce qui sort du séquenceur sont des séquences d’ADN de quelques centaines de base dont on ne sait rien : ni comment elles sont ordonnées, dans quel sens elles ont été séquencées (pour faire simple, dans le sens montant ou descendant de l’ADN…), d’autant plus qu’elles se recouvrent les unes avec les autres, ni où elles sont positionnées dans l’ADN et encore moins à quel chromosome elles correspondent. De plus, la tâche est compliquée par les 1% à 2% d’erreurs générés par les séquenceurs. Sans compter le fait que dans les séquences dites “non codantes” qui constituent l’essentiel de l’ADN, il existe une foison de grands blocs d’ADN dupliqués.
L’alignement va être la première étape de reconstitution du puzzle : il consiste à trouver comment les brins séquencés s’alignent les uns par rapport aux autres, en exploitant leurs recouvrements. Si le séquençage est “di novo”, le travail est très lourd. Si l’on peut exploiter un génome de référence, l’alignement sera plus facile.
L’alignement créé ce que l’on appelle des “contig”, des séquences continues de bases. Il va manipuler beaucoup de données (100 Go !) et nécessiter énormément de calculs. Ceux-ci sont réalisés soit en local, soit, ce qui est de plus en plus courant “dans le cloud”. Mais il faut du très haut débit pour uploader le résultat du séquençage dans le cloud. D’où le fait que les laboratoires de recherche sont souvent connectés à Internet par des liaisons spécialisées très haut débit (plus de 1 Gbits/s). Dans certains cas, on va jusqu’à envoyer un disque dur aux services de cloud ! Cela reste encore un peu artisanal (source).
Le principal logiciel pour l’alignement des séquences est BLAST (Basic Local Alignment Tool), créé en 1990 et qui utilise aussi l’algorithme de Smith-Waterman de “programmation dynamique”. Son équivalent dans le cloud est … CloudBLAST. On peut aussi citer Bowtie(Ultrafast Short Read Aligner, très économe en mémoire, et est lui-même utilisé dans un tas d’autres logiciels), MAQ (qui est plus lent et donc bien moins intéressant que Bowtie), Mosaik (qui utilise l’algorithme de Smith-Waterman) et Novoalign (pour Linux et MacOS). Ces logiciels sont soit open source, soit gratuits à l’usage pour les activités non lucratives et de recherche. Il y en a en fait des dizaines.
Ces logiciels s’appuient sur des briques techniques comme MapReduce, un framework distribué créé par Google qui permet de paralléliser les traitements, le framework Java Hadoop qui fait partie des travaux de l’Apache Foundation, OSS version, le HDFS (Hadoop Distributed File System), CloudBurst qui permet de créer un index de k-mer (suites de bases) pour identifier leur alignement et qui utilise lui-même MapReduce ou encore MUMerGPU, qui parallélise le calcul sur des GPU comme ceux de nVidia. Sachant que l’outil date de 2007 et les GPU ont fait d’énormes progrès depuis et peuvent maintenant intégrer plus d’un millier de cœurs. Voici une présentation qui décrit les algorithmes utilisés pour l’alignement.
- Cartographie
Cette étape va répartir les séquences alignées par région (le plus souvent, les gènes) et par chromosome. Elle va faire appel aux nombreuses bases de données de gènes, cf la partie qui concerne les bases de données un peu plus loin. Techniquement, cette cartographie repose aussi sur des logiciels d’alignement. Au lieu d’aligner les séquences d’ADN sorties d’un séquenceur, on aligne les “contigs” (résultat d’alignement de séquençage) avec des séquences de référence stockées dans des bases de données. Là encore, les algorithmes associés ont besoin de pas mal de puissance de calcul.
- Réduction
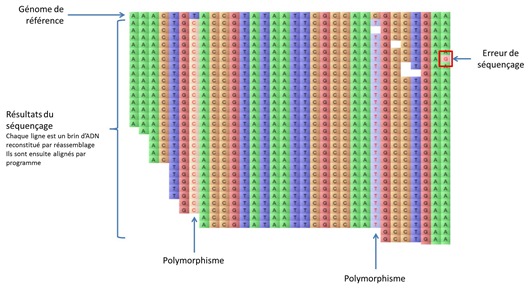
Il s’agit de la phase d’identification des erreurs de séquençage et des zones de polymorphisme (SNP). Le schéma ci-dessous illustre ce travail de manière visuelle : en haut nous avons un génome de référence de l’ADN étudié et en dessous une trentaine de séquences réassemblées par le logiciel. Cela permet d’identifier ici deux polymorphismes, une base modifiée par rapport à l’ADN de référence, et une erreur de séquençage, facilement éliminée statistiquement.
L’offre logicielle associée à ces quatre étapes est énorme, avec principalement de l’open source, ce qui s’explique par le fait qu’ils sont avant tout utilisés par des laboratoires de recherche. On trouve à la fois des logiciels traitant des briques de base comme l’alignement ou des logiciels intégrant plusieurs briques et simplifiant le processus pour les utilisateurs.
Dans le cloud, il y a notamment CrossBow qui permet d’identifier des SNPs. Il exploite une implémentation Hadoop du logiciel MapReduce pour exécuter en parallèle plusieurs instances du logiciel d’alignement Bowtie (je l’avais survolé dans un article sur les Googles Labs en 2006). Il utilise par ailleurs plusieurs instances du logiciel d’identification de polymorphismes SOAPsnp qui fonctionne avec un algorithme bayésien. Crossbow est capable de traiter l’ADN complet d’une personne en quatre heures, ceci comprenant le temps d’upload. Et pour $85. La recherche de SNPs coutait $100 en 2009 sur Amazon EC2. Il y a aussi SOAPdenovo, qui est capable d’assembler un génome humain complet “de novo” avec des données issues d’un séquenceur Illumina avec un taux de couverture de 52. Il faut toutefois 1500 heures de CPU sur un machine à 32 cœurs (soient 2 jours) et la bagatelle de 512 Go de RAM. On peut enfin citer DNAnexus, une startup dans laquelle a investi Google Ventures qui fournit des services de traitement de données de séquençage en cloud.
- Visualisation et analyse
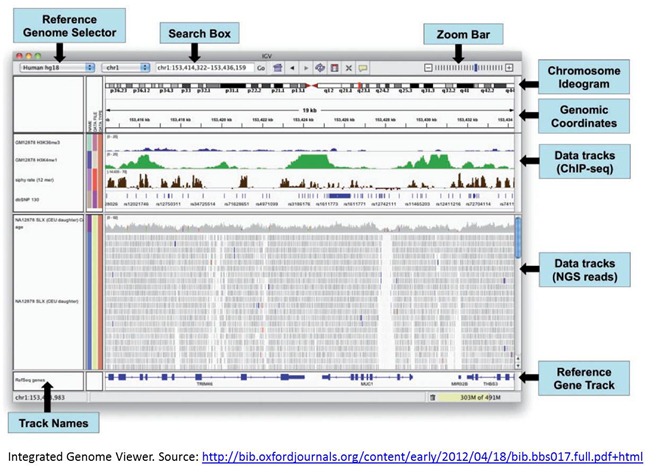
Une fois les basiques réalisés, la visualisation des résultats et leur croisement avec des bases diverses, notamment de gènes, donne lieu à beaucoup de créativité et d’amélioration de la productivité pour les chercheurs. Les outils de navigation dans les génomes fonctionnent soit en ligne soit dans des applications locales. Dans ce dernier cas, il s’agit souvent d’applications écrites en Java, ce qui permet d’en avoir une version “en ligne” – en fait, fonctionnant dans un navigateur mais cela reste du “client lourd” – avec le même code. Voici par exemple l’Integrated Genomic Viewer (ci-dessous).
Citons le projet Open Genomics Engine, sponsorisé par nVidia qui rappelle l’utilité d’aller vite dans le séquençage du génome en évoquant comme exemple l’épidémie d’Escherichia Coli génératrice de toxine qui avait sévi en Allemagne pendant l’été 2011 et qui avait tué 17 personnes. Le gène responsable avait été séquencé en trois heures avec une machine Ion Torrent (décrite dans l’article précédent de cette série) ce qui permis d’en détecter l’origine avec précision (une production de graines germées en Allemagne) et puis de la stopper.
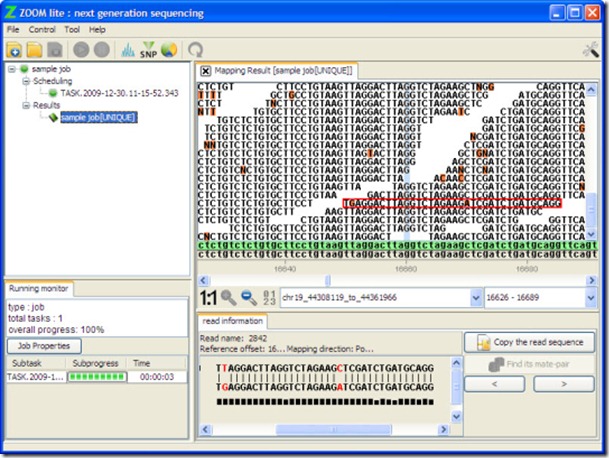
Les logiciels ne manquent pas dans cette catégorie. On peut aussi évoquer GenPlay, qui est écrit en Java et exploite les données de séquençage de toutes sortes (ADN, ARN, ChIP-Seq, TimEX-Seq, SNP), Savant Genome Browser, Magic Viewer, GenomeView ou encore Zoom Lite qui se télécharge sur CNET (ci-dessous) !
Comment faire son marché ? Il faut tester une bonne douzaine d’outils pour faire son choix, et probablement s’abonner à la littérature technique du domaine.
Bases de données
Quid du format des séquences d’ADN générées par les séquenceurs et enregistrés dans les bases de données ? Il existe tout un tas de formats qui vont des formats bruts de décoffrage à des formats contenant des annotations et tags divers. Il y a par exemple le format texte FASTA qui occupe un octet par base et nécessite donc 3 Go par génome humain ou 6 Go dans sa forme diploïde (chromosomes doubles séquencés). Il existe aussi un format dense qui code les bases sur 2 bits. Il est exploité par certains logiciels de visualisation d’ADN. Vous pouvez même par exemple télécharger les 700 Mo de l’ADN du chromosome 18 humain si cela vous chante. Et vous vous demanderez comme moi pourquoi les 76 millions de bases de ce chromosome occupent chacune près de 9 octets dans ce format qui pourtant ne contient pas d’annotations
Côté bases de données, il y a l’embarras du choix selon les besoins. On trouve des bases sur les génomes d’espèces diverses, sur les transcriptomes (ADN codantes), sur les protéines (y compris leur modèle 3D), sur l’ADN des bactéries, sur les gènes du cancer, sur les protéines, sur les ribosomes, sur l’effet des médicaments, etc. Et ces bases sont de plus en plus croisées.
Les bases de données dans le domaine sont très ouvertes et accessibles à tous. Il faut bien entendu avoir un minimum de compétences (que je n’ai pas) pour pouvoir exploiter tous ces outils. Mais le fait est que la bio-informatique est une discipline où le data-mining est librement accessible. Cela vient du fait que le vivant ne peut pas se breveter, notamment depuis que l’ADN fait partie du patrimoine de l’humanité selon la déclaration de l’UNESCO du 11 novembre 1997. Un gène n’est pas brevetable non plus en Europe… mais peut l’être aux USA. Les thérapies ou des modifications du vivant peuvent aussi l’être, comme l’invention d’un procédé de fabrication d’une protéine. En tout cas, les séquences d’ADN “naturel” des bases de données sont librement accessibles et exploitables (cf cet excellent document sur la brevetabilité du vivant). Nous sommes ici dans un environnement “d’open data”.
La base de référence sur le génome humain est la Genbank, du National Center for Biotechnology Information, encore une fois, financée par le NIH américain. La base est mise à jour tous les deux mois. En aout 2012, elle comportait 143 milliards de bases d’une quinzaine d’espèces différentes, dont 16 sur nous, les homo sapiens. La Genbank américaine a son pendant européen, l’European Nucleotide Archive (ENA) et japonais, la DNA Data Bank of Japan, les trois mutualisant leurs bases et collaborant au sein de la International Nucleotide Sequence Database Collaboration (INSDC) sur la standardisation des formats de données. A noter le rôle dans l’histoire de l’EMBL, l’European Molecular Biology Laboratory qui est basé à Grenoble et qui se focalise sur des projets de recherche en épigénétique et s’appuie sur des moyens d’investigation de pointe (diffraction par rayons X, etc).
Les données de Genbank sont accessibles via l’outil BLAST qui contient des séquences de nucléotides qui ont été assemblées avec l’outil du même nom. Les bases de nucléotides se focalisent sur le cDNA (coding DNA), soit l’ADN codante des gènes et non l’ADN non codante qui l’entoure et qui relève de plus de 99% de l’ADN du génome humain. BLAST permet également d’accéder à des bases de protéines.
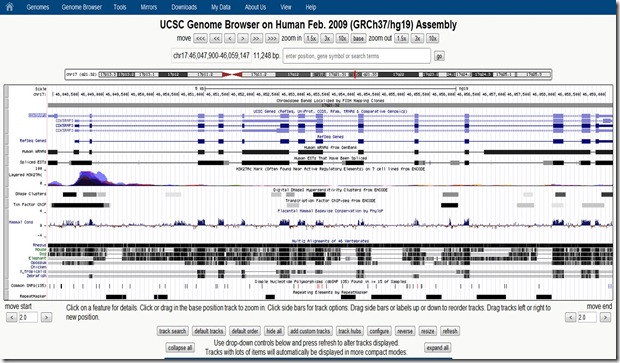
Il faut aussi citer la base Ensembl, qui permet de naviguer dans les génomes de nombreux organismes. La base est non seulement librement accessible mais également exposée sous forme d’APIs. Et puis l’UCSC Genome Browser. Ces outils visualisent aussi les annotations réalisées par tous les chercheurs à l’échelle mondiale, qui les sont publiées de manière ouverte. Cela permet de bien mutualiser le savoir sur les gènes et leur expression. Le lien croisé entre les bases semble réalisé au coup par coup, mais il semble que les outils de dernière génération relèvent de plus en plus du data-mining multi-bases et soient de plus en plus puissants pour exploiter et croiser des sources de données diverses. Les bases sont aussi de plus en plus croisées avec les bases de données référençant les publications scientifiques comme PubMed – encore une autre branche du NIH – sachant que certains articles sont d’accès payant.
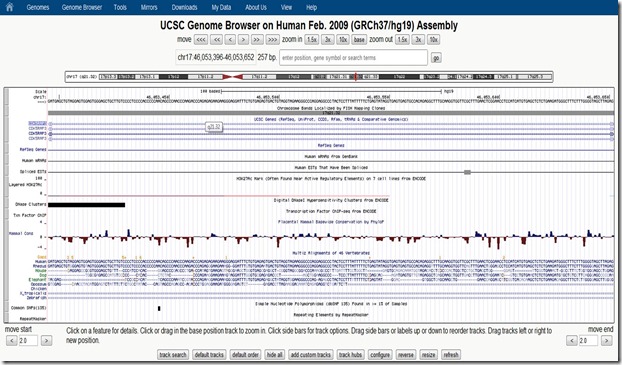
Petit parcours rapide avec le Genome Browser : on peut visualiser à haut niveau la cartographie d’un chromosome à l’endroit d’un gène que l’on veut étudier. D’un coup d’œil (ci-dessus), on peut identifier la position du gène (en utilisant son code) et en zoomant, les séquences d’ADN correspondantes (ci-dessous) non seulement pour l’homme mais pour un tas d’espèces animales ainsi que les polymorphismes identifiés.
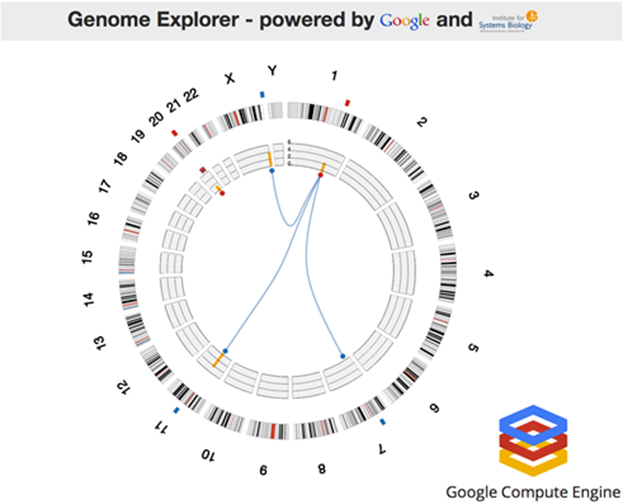
Faisons maintenant un tour dans le Genome Explorer qui avait donné lieu à une impressionnante démonstration lors de la dernière conférence développeur Google I/O (juin 2012, vidéo de 4 minutes). La démonstration s’appuyait sur Google Compute Engine et une application de l’Institute for Systems Biology qui tournait sur 10000 cœurs et 1024 serveurs octocœurs. Le processus s’exécute en une heure. La démonstration était ensuite étendue à 600000 cœurs (sur 771000 disponibles) pour s’exécuter plus rapidement… en quelques secondes et sous les yeux zébahis de l’assistance. Le logiciel exploitait le RF-ACE code, un algorithme d’auto-apprentissage qui identifie les associations entre caractéristiques génomiques. L’ISB est un laboratoire de recherche privé à but non lucratif basé à Seattle qui est notamment très pointu en exploitation de techniques de bio-informatique (spécialité de 10 de ses 23 top chercheurs). Google permet à une douzaine de laboratoires dans le monde d’exploiter à cette échelle la puissance de son Compute Engine dans le cloud.
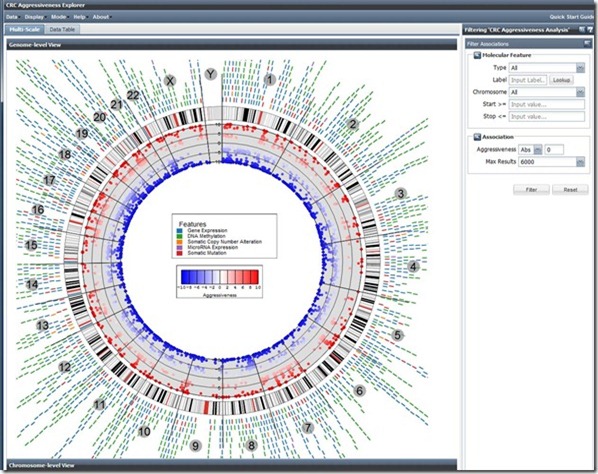
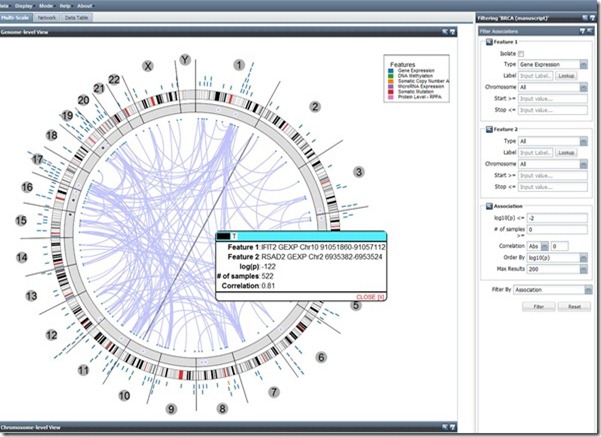
L’application scientifique ? Il s’agissait d’une visualisation du Cancer Regulome créé dans le cadre du projet The Cancer Genome Atlas qui vise à cartographier les gènes du cancer ainsi que les facteurs externes provocant leur expression. La cartographie présentait relevait d’une analyse multivariante faisant la corrélation entre les gènes, les mutations des gènes et les données cliniques (personnes affectées ou non de cancers de types variés). Ce type d’analyse a notamment permis d’identifier des gènes co-responsables de cancers colo-rectaux, avec à la clé, la création de thérapies ciblées pour combattre ces cancers à la source.
Basée sur les mêmes technologies de Google, la cartographie ci-dessous obtenue dans le Cancer Regulome consolide quant à elle les informations sur les gènes du cancer et les facteurs épigénétiques d’expression des gènes comme la méthylation de l’ADN ainsi que les mutations d’origines diverses.
Dans ce second écran, on peut visualiser d’un coup d’œil les relations entre gènes de l’ensemble des chromosomes (ne m’en demandez pas plus…).
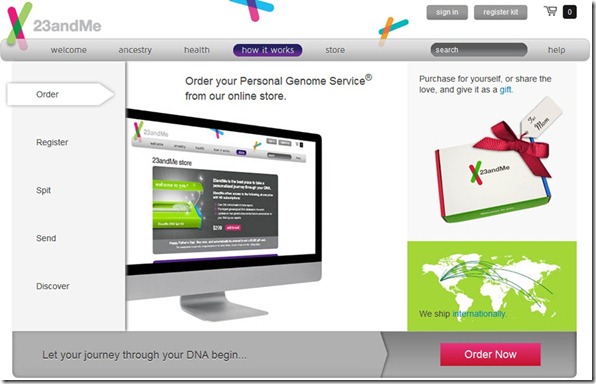
Pourquoi Google s’intéresse-t-il à la génétique ? Certains patrons du CAC 40 s’intéressent à l’art (Pinault), à la voile, ou à d’autres passions. Ici, la motivation est aussi très personnelle à la source. Tout d’abord la femme de Sergei Brin, Anne Wojcicki, est cofondatrice de la startup 23andMe (Mountain View, $52m de levés), dans laquelle Google Ventures et Sergei Brin ont investit. Secundo, Sergei Brin a appris par la seconde qu’il était atteint d’une mutation génétique génératrice de la maladie de Parkinson. Pour couronner le tout, Lucy Page née Southworth, la femme de Larry Page, l’autre cofondateur de Google, est docteur en bio-informatique de Stanford. Ceci explique pourquoi Google finance le XPrize dont nous avons déjà parlé !
23andMe permet au grand public d’analyser son génome pour identifier ses origines, ses ancêtres et ses potentialités de pathologies d’origine génétique (diabète type 2, maladie de Parkinson, etc) mais aussi certaines de vos allergies alimentaires et les effets indésirables de certains médicaments. Il propose aussi aux utilisateurs de créer leur propre réseau social avec leurs parents et cousins plus ou moins éloignés, pour identifier les traits qu’ils ont en commun. Des données que Google se ferait un plaisir d’indexer ! En bonus, on vous indiquera votre variante du gène ACTN3 qui produit la protéine Alpha-actinie-3, celle qui conditionne la performance athlétique, au sprint ou à l’endurance (mais pas les deux à la fois). Le tout pour $300 ! Le processus : ils envoient un petit kit permettant de leur envoyer un échantillon de salive et en trois semaines, on obtient le résultat dans un mail sécurisé. Est-ce une lubie d’hypochondriaque ? Comme tous les outils un peu génériques, on peut en faire n’importe quoi, du meilleur au pire. Mais il semble qu’il sera difficile d’arrêter cette tendance tout du moins aux USA.
Ce genre de service s’appuie sur une technologie qui n’est pas celle du séquençage. 23andme utilise le système Illumina OmniExpress Plus Genotyping BeadChip qui sert à identifier les variations de notre ADN (“SNP” : single nucleotide polymorphisms) avec des biopuces. Dans la machines d’Illumina, ce sont des centaines de milliers de variations qui sont tout de même identifiables et le traitement est réalisé en quelques minutes. C’est bien plus rapide qu’un séquençage d’ADN. Il se trouve que le logiciel d’analyse d’Illumina (Genome Studio) dispose d’API ouvertes exploitables par des tiers. C’est peut-être ce que 23andMe utilise pour alimenter ses propres bases et son service en ligne pour ses clients.
Pour terminer ici, un petit mot sur une technologie dérivée de celle du séquençage : la technique inverse consistant à stocker de l’information dans de l’ADN. Une équipe de Harvard a réussi à le faire. L’équipe de George Church and Sri Kosuri du Wyss Institute aurait réussi à stocker 700 To de données dans un gramme d’ADN. Le tout avec des machines de laboratoire du marché.
Le système encode les données sur des brins d’ADN courts de 96 bases, chaque base représentant un bit (1 pour une paire T-G et 0 pour une paire A-C, quelle que soit l’orientation du couple, T-G ou G-T sur le double brin d’ADN). Chaque bloc de 96 bases comprend un bloc d’adressage de 19 bits. Vous me direz que cela ne fait que 2 puissance 19 combinaisons, soit 524288. C’était suffisant pour le test des chercheurs qui ont utilisé 54898 blocs de 96 bits pour stocker un bouquin (à eux) de 528 Ko. Ils ont reproduit le test plusieurs fois, ce qui leur a permis de générer 700 To de données. Mais avec beaucoup de redondance. Pour ne pas avoir de limites, il serait bon de partir tout de suite avec un bloc d’adresse de 128 bits, comme pour TCP/IP V6. Et là, il faudrait donc avoir des blocs d’ADN d’au moins 256 bases voire plus. Ce qui compliquerait un peu la tâche du séquençage.
L’avantage de l’ADN dans tout ça ? Il peut se conserver très durablement, tout du moins dans l’Ethanol, et bien plus durablement qu’un disque magnétique ou même qu’un DVD qui s’use par érosion chimique à l’échelle d’une ou deux décennies, bien que l’on manque encore de recul. Les inconvénients ? Cela reste du stockage de long terme et dont le temps d’accès restera encore longtemps très insatisfaisant.
Les chercheurs précisent bien que l’ADN généré pour stocker l’information n’est pas placé dans des cellules vivantes. Celles-ci auraient vite fait de faire évoluer l’ADN par mutations et de modifier l’information stockée. Un inconvénient bien pratique qui permet au passage d’évacuer les les risques “sanitaires” que cela pourrait générer. Notamment au niveau de l’infection de l’ADN par des virus informatiques qui deviendraient des virus vivants.
Bref, l’ADN, ou plutôt ce que l’homme en fait, n’a pas fini de nous surprendre.
Dans l’épisode suivant, nous sortirons un peu du cadre purement technologique pour observer d’où viennent toutes ces innovations. Pourquoi viennent-elles essentiellement des USA. Très peu d’Europe. Et aucune de France !
![]()
![]()
![]()
Reçevez par email les alertes de parution de nouveaux articles :
![]()
![]()
![]()































 Articles
Articles
Stocker des données dans de l’ADN? Il fallait y penser… A quand la prise USB sur l’oreille du patient
Bonjour, merci pour vôtre article très complet, c’est très instéressant, mais je ne suis pas trop daccord quand vous dites que le séquençage pour tous est un simple hobbit de la part des fondateurs de google ( avec 23 and me ), moi je crois que c’est un investissement très intéligent et bien réfléchie comme tous ceux que font cette firme.
Grâce à #TVdebout j’ai appris l’existence de la technlogie du génome. https://t.co/ClAQxM4S4r crtl+f puis Genome Explorer
En (un) sept mot(s)
Bravo pour cet article !