Depuis quelques années, je profite de l’été pour creuser un sujet un peu hors des sentiers battus. En 2009, c’était sur la capture d’images d’Apollo 11 à l’occasion du quarantième anniversaire des premiers pas de l’homme sur la Lune. L’année dernière, c’était sur les racines anciennes du retard français dans l’adoption des technologies de la communication. Cette année, changement de braquet. On passe à la génétique ! Ce sont un peu mes “devoirs de vacances”. Chacun son truc… !
Dans le Rapport du Consumer Electronics Show de Las Vegas de 2012, j’avais rapidement évoqué le cas d’une machine permettant le séquençage du génome humain de manière relativement abordable et en une journée :
“Enfin, on pouvait voir ce Proton de la société Ion Torrent, un analyseur d’ADN (semiconductor sequencer) sur le stand de Scientific American. Ce séquenceur ADN décode votre génome en une journée et pour $1000 avec tout cela comporte comme implication dans la détection de pathologies et terrain génétique favorable ou pas. Il fonctionne avec un capteur CMOS voisin de celui d’un appareil photo capable de décoder 660 millions de génomes en une journée. Comment ça marche ? Assez compliqué à comprendre et à expliquer. Ces capteurs CMOS ont des transistors à effet de champs qui détectent la quantité d’ions d’hydrogènes générés par la polymérisation de l’ADN. On peut mettre un peu plus d’un million de ces transistors sur ces capteurs CMOS. La machine est vendue $150K. Comme l’impression 3D, encore un truc qui va devenir mainstream dans peu de temps ! Littérature sur la question ici et là. Avec les technologies de big data, ce n’est rien de moins que la guérison des cancers grâce à l’analyse génétique et à des traitements ciblés qui est anticipée”.
J’ai voulu en savoir plus pour comprendre comment ce genre d’engin pouvait fonctionner. C’est bien le tout d’évoquer un capteur CMOS et ses millions de pixels. Mais là, ce n’est pas de la photo. Il s’agit d’identifier les centaines de millions de séquences de nucléobases qui s’enchainent dans l’ADN de nos chromosomes. Conceptuellement, c’est assez complexe, et bien plus que la mécanique de détection des fameux bosons de Higgs qui repose sur l’envoi les uns contre les autres de protons ultra-énergisés dans l’énorme LHC du CERN de Genève.
Comment ces pixels de CMOS font-ils donc pour détecter les séquences d’ADN ? Comment la machine les met-elle dans l’ordre ? Quelle différence il y-a-t-il entre le séquençage générique du génome humain et celui, personnalisé, de tout un chacun ? Quelles pathologies peut-on traiter grâce à un séquençage pour tous ? Comment ces technologies vont-elles ou peuvent-elles se commoditiser ? A quel prix ? Et avec quels types de machines ?
Quel est donc le lien avec la ligne éditoriale de ce blog ? La biologie y est clairement hors-sujet. Elle pourrait même nous emmener loin dans les méandres des débats entre évolutionnistes et créationnistes. En effet, la biologie moléculaire est incroyablement complexe et riche de molécules et processus chimiques divers. Quand on les décortique un par un, on leur trouve tous une causalité scientifique et leur origine peut souvent s’expliquer par des processus de nature évolutionniste. Mais on peut facilement être fasciné par le génie du vivant et lui chercher un “créateur”. D’où les théories du “design intelligent” largement débattues aux USA.
On va tout de même revenir à la technologie “de base” avec les capteurs CMOS et les traitements numériques de l’information. Il y aura même des morceaux de big data dedans, même si ce n’est pas ma tasse de thé. Et on parlera aussi d’initiatives d’IBM et de Google dans le domaine.
Alors, allons-y ! Cela nécessitera plusieurs parties : d’abord, un rappel des basiques de l’ADN façon cours de sciences naturelles du secondaire, puis sur les techniques de son séquencement et enfin, un tour dans la partie numérique des opérations.
Décortiquer l’ADN
L’ADN, tout le monde en a une idée générale. On en a tous entendu parler à l’école. C’est une molécule qui contient le code génétique à l’origine de la vie animale et végétale, qui permet la reproduction mais qui explique aussi certaines pathologies dites génétiquement transmissibles.
Avant de voir comment on décode l’ADN, on va tout de même reprendre les basiques de la biologie moléculaire pour établir le vocabulaire, assez riche, du sujet. Nous allons pour ce faire emboiter une à une les poupées russes du vivant en allant du plus petit au plus grand, sachant que je me suis très largement appuyé sur Wikipedia pour reconstituer cette série, mais pas seulement, ayant trouvé pas mal de littérature sur le sujet dont un article fondamental publié en février 2001 dans Nature : “Initial sequencing and analysis of the human genome”. Je vous épargne tout de même l’échelle subatomique !
Quand j’en trouve la trace, j’indique la date et le ou les auteurs de la découverte de la structure en question, qui sont très souvent devenus des prix Nobel de médecine. Je fais aussi le parallèle côté dimensions entre le code génétique et les micro-processeurs d’aujourd’hui, histoire de comprendre le défi qui se pose dans le décodage du génome. A vrai dire, l’écriture de cette série d’articles m’a fait découvrir un monde fascinant de connaissances, qui se développe de manière exponentielle depuis des décennies. La connaissance que l’on a des mécanismes du vivant est incroyablement détaillée, et en même temps toujours insatisfaisante au vu de la difficulté à trouver des traitements adéquats à certaines pathologies (myopathies, cancers, diabète, maladies tropicales, etc).
Dans nous allons examiner dans l’ordre croissant d’intégration les éléments suivants : nucléobases, nucléotides, ADN, codons, séquences codantes, gènes, nucléosomes, nucléosomes, nucléofilaments, chromatine, chromosome, nucléole avec un petit détour par les mitochondries, noyau, génome et enfin cellule. C’est parti…
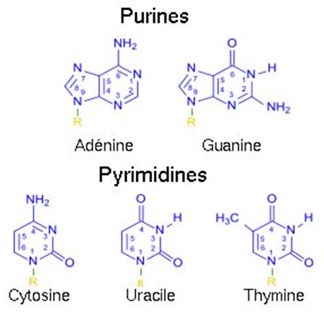
Nucléobases
Ce sont les molécules de base de la construction de l’ADN et de l’ARN qui est créé à partir de l’ADN. Il y en a cinq différentes : l’adénine (A), la cytosine (C), la guanine (G), la thymine (T) et l’uracile (U). Les quatre premières se trouvent dans l’ADN et la dernière se trouve dans les différentes formes d’ARN, en lieu et place de la thymine. Ces molécules sont à base d’azote, d’hydrogène, d’oxygène et de carbone. L’identification de ces molécules date de 1929 par le russo-américain Phoebus Aaron Levene.
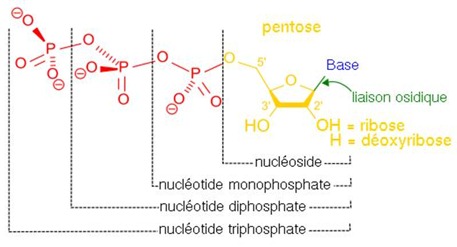
Nucléotides
Ce sont des molécules organiques qui constituent les brins d’ADN. Elles s’appuient sur une des quatre nucléobases de l’ADN, un sucre (un pentose, soit une molécule monosaccaride dotée de cinq atomes de carbone) et des groupements phosphatés (mono, di ou triphosphates).
Plus précisément, l’ADN s’appuie sur des nucléotides monophosphatées : le dAMP (désoxyadénosine monophosphate), le dTMP (désoxythymidine monophosphate), le dGMP (désoxyguanosine monophosphate) et le dCMP (désoxycytosine monophosphate). Mais lorsque l’on parle du décodage de l’ADN, on utilise comme système de notation celui des nucléobases (A, C, G et T) que contiennent ces nucléotides (dAMP, dCMP, dGMP et dTMP). Les chromosomes humains comprennent près de 6 milliards de nucléotides.
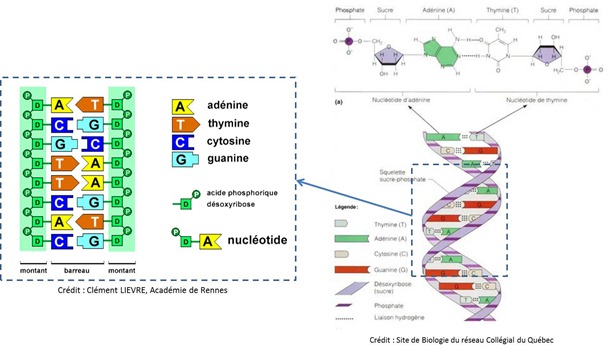
ADN
Acide désoxyribonucléique, c’est la longue chaine qui contient le code génétique dans les chromosomes de nos cellules. Sa structure moléculaire de base est en double hélice qui comprend une suite de paires de bases entourées de leur sucre-phosphate qui sont reliés entre elles par des liaisons sucre-phosphate.
Côté bases, chaque côté de l’hélice est le miroir de l’autre : une base A est toujours associée à une base T (via deux liaisons hydrogène) et une base G à une C (via trois liaisons hydrogène). Cet agencement a été découvert grâce à une technique de diffraction aux rayons X mise en œuvre en premier sur l’ADN par Rosalind Franklin en 1952. Il s’appuyait sur la découverte antérieure, de Chargaff, en 1949, de la proportion équivalente des bases A et T puis G et C dans les cellules, et constante pour chaque espèce vivante. Les liaisons hydrogène – qui relient les paires de bases dans l’ADN – sont de faible énergie, ce qui facilite la séparation des brins d’ADN et leur réplication. On peut ainsi séparer les deux brins d’un ADN par simple réchauffement alors que celui-ci ne séparera pas les nucléotides les uns des autres car ils sont reliés par une liaison de forte énergie (oxygène-phosphate).
Les deux hélices de l’ADN ne sont pas espacées de manière égalée : le grand sillon fait 2,2 nm de haut et le petit, 1,2 nm. Comme tout s’explique, cela vient de l’angle des liaisons hydrogène qui associent les paires de bases. Un peu comme si on tirait vers le haut ou vers le bas une des hélices de l’ADN.
C’est en 1958 que Meselson et Stahl découvrent le processus de réplications dite semi-conservative de l’ADN qui voit chaque bras de l’hélice répliqué pour créer une molécule d’ADN identique à celle d’origine. Sachant néanmoins que l’on distingue toujours un bras “original” et un bras contenant une sorte de négatif, un peu comme dans la photo argentique. Le bras original est identifié par le sens des liaisons phosphate-sucre entre les nucléotides.
Il y a environ deux mètres linéaires d’ADN dans chaque cellule humaine sachant que l’ADN y est très dense comme nous allons le voir plus loin. La découverte de L’existence de l’ADN et de son fonctionnement a pris plus d’un siècle avec pour commencer son identification en 1869 par Miescher et la modélisation de sa structure en hélice et paires de bases par Watson et Crick en 1953. Le fonctionnement de la machinerie de la reproduction des cellules ainsi que celle de la création des protéines ont été découvertes dans les décennies suivant 1953.
Codons
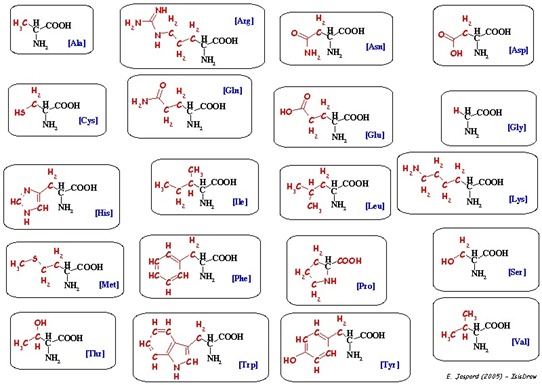
Identifiés par Brenner et Crick en 1960 puis Nirenberg et Matthaei en 1961, il s’agit du niveau d’intégration suivant dans l’ADN et la chaine du vivant. Ce sont des séquences de trois nucléotides spécifiant l’un des 22 acides aminés qui sont eux-mêmes les primitives de constitution des protéines qui servent de base au fonctionnement interne des cellules vivantes. Les acides aminés ont été découverts entre le début et la fin du 19ième siècle. D’où viennent-ils dans le corps humain ? 12 sont synthétisés par le processus métabolique par découpage des protéines de notre alimentation et 9 sont d’origine externe.
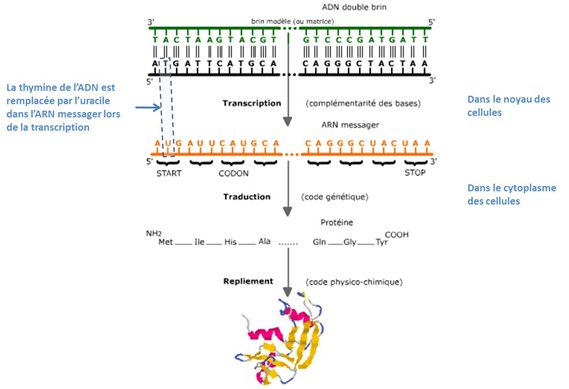
Ces séquences de codons se trouvent dans l’ADN. Elles sont transmises à de l’ARNm (acide ribonucléique messager) lors de la transcription des gènes de l’ADN qui s’appuie sur de l’ARN polymérase. L’ARNm sort du noyau des cellules pour atteindre le cytoplasme où il est transformé en protéines grâce à l’action de ribosomes, des protéines complexes et de l’ARNt (ARN de transfert).
La composition de l’ARN a été découverte par Volkin et Astrachan en 1956. Le rôle de l’ARNm a été identifié en 1961 par les français Monod et Jacob. Autre manière de décrire cette belle mécanique : l’ARNm est un code qui est exploité par les outils que sont les ribosomes et l’ARNt pour créer les protéines.
La succession des codons sur l’ARNm détermine la structure primaire de la protéine qui sera synthétisée, soit l’enchainement linéaire des molécules. La “structure secondaire” décrit son organisation tridimensionnelle et la “structure tertiaire” décrit la manière dont la molécule se replie sur elle-même, ce qui donne à la protéine sa fonctionnalité. Ces repliements sont la conséquence physique et chimique de la composition en acides aminés des protéines. Dans certains cas se créent des sites actifs que l’on appelle les enzymes.
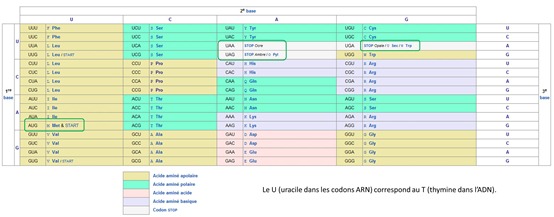
Les ARN messagers créés par transcription de l’ADN sont constitués d’une succession de plusieurs dizaines à centaines de nucléotides. Nous avons vu que dans l’ARN, quatre bases nucléiques déterminent la séquence d’un codon : adénine, guanine, uracile et cytosine. Ce qui donne 43 = 64 codons différents, servant au codage de 22 acides aminés différents (formules chimiques ci-dessous ).
La table de correspondance ci-dessous a été identifiée en 1961 par Nirenberg. Un codon “start” (ATG en notation ADN ou AUG en notation ARN) commande le départ de la synthèse des protéines et trois codons “stop” en commandent l’arrêt. Mais il existe une séquence préalable au codon start qui annonce les gènes : le “promoteur”. C’est sur lui que se fixe l’ARN polymérase qui va déclencher la copie de l’ADN en ARNm. Un peu comme le début d’une fermeture éclair.
Mais d’où viennent ces ribosomes qui utilisent l’ARNm et les acides aminés pour construire les protéines ? Pour résoudre le problème de la poule et de l’œuf, les molécules de ribosomes sont elles-mêmes construites à partir d’ARNr (ARN ribosomique) obtenu par transfert de gènes de l’ADN. Mais le processus de leur création est assez complexe car l’ARNr est obtenu via une pré-ARNr qui est découpée en trois ARNr.
Les 52 protéines constitutives des ribosomes sont quant à elles produites par le cycle normal via de l’ARNm et d’autres ribosomes. Ce sont ces protéines qui sont associées avec les les ARN ribosomiques pour créer des pré-ribosomes. Enfin, cela se termine par un processus de maturation. Le tout dans différentes parties des cellules. Bon, et les 52 protéines constitutives des ribosomes, elles viennent d’où ? Elles aussi de la transcription d’ADN en ARNm et de leur utilisation pour l’assemblage d’acides aminés… par d’autres ribosomes !
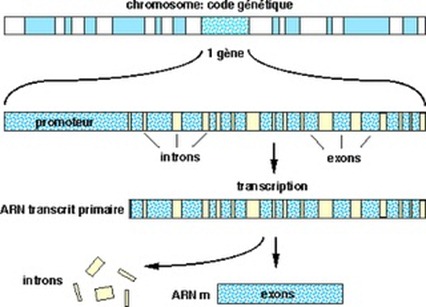
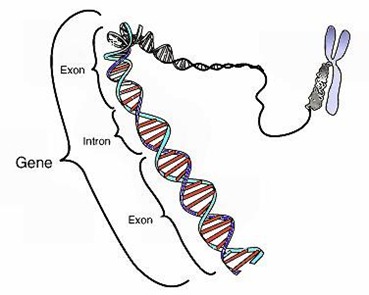
Séquence codante (CDS ou Coding DNA Sequence, aussi simplifié en cDNA)
Partie d’un gène qui, après avoir été transcrite en ARNm dans le noyau des cellules, est traduite en protéines. Les gènes sont en effet constitués dans l’ADN de suites alternant des séquences codantes (exons) et des séquences non codantes (introns, découverts en 1993). Les exons commencent par le codon ATG (trois nucléotides à bases adénine-thymine-guanine) et se terminent par un codon stop (TAA, TAG, ou TGA). Le processus de création des protéines via l’ARNm implique une mécanique d’élimination des introns qui ne servent à rien que l’on dénomme l’épissage et qui est lui-même un mécanisme très compliqué. Les gènes occupent environ 0,5% de l’ADN humain et les séquences codantes des gènes n’en représentent qu’environ 5%, soit 0,025% de nos chromosomes. Ce qui ne veut pas dire pour autant que le reste ne sert à rien, mais le niveau de connaissance est moins bon sur la partie “hors gènes” de l’ADN que dans les gènes.
Gène
C’est l’unité de base d’information génétique qui se présente sous la forme d’une séquence d’ADN qui spécifie la synthèse de chaînes de polypeptides (chaînes de 10 à 100 acides aminés reliés par des liaisons peptidiques) qui servent elles-mêmes à la génération de protéines, qui sont des polypeptides “longs” (1941, Beadle et Tatum puis 1944-1946, Avery). Mais leur activation (“expression”) dépend du type des cellules. L’identification de la série de bases qui démarrent et terminent un gène date des années 1970. L’ADN humain comprendrait environ 23000 gènes selon les connaissances à ce jour. Les estimations du nombre de gènes ont été très variables ces 50 dernières années. Elle allaient jusqu’à 100000 mais leur nombre s’est ensuite réduit après le séquençage complet de l’ADN humain terminé au début des années 2000. Ce n’est pas le tout de séquencer le génome, il faut comprendre à quoi servent les séquences d’ADN !
Dans chaque cellule humaine, il y a environ 10000 gènes qui sont exprimés et représentent les fonctions communes de vie des cellules. Et environ 1000 gènes sont exprimés qui sont liés au type de la cellule : musculaire, nerveuse, osseuse, sanguine, etc.
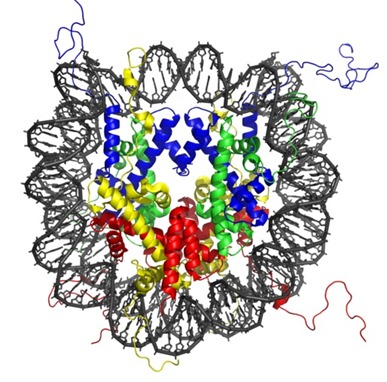
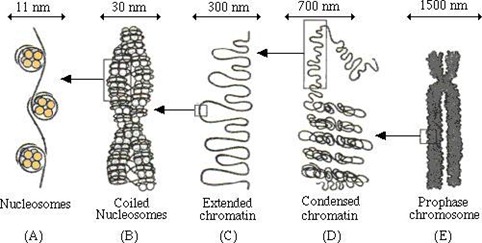
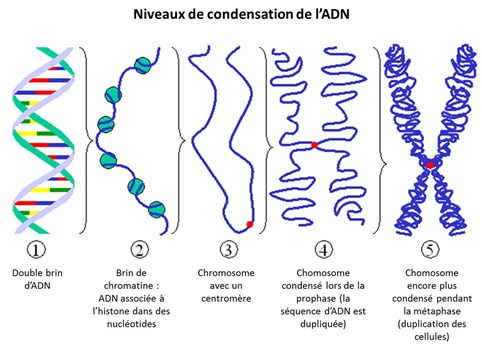
Nucléosomes
C’est un complexe d’ADN et de protéines qui constitue une unité de base de la chromatine que nous verrons plus loin. C’est le premier niveau de compaction de l’ADN dans les chromosomes. Le nucléosome est un enroulement d’environ 140 paires de bases d’ADN autour de protéines complexes dont les histones (en couleur ci-dessous) qui sont très riches en acides aminés basiques. Il y a deux sortes d’histones, les 2A, 2B 3 et 4 autour desquelles l’ADN s’enroule et l’histone 1 qui se place à l’extérieur de l’ensemble, comme un verrou.
Un nucléosome fait environ 11nm de diamètre. Mais la partie de l’ADN qui est ainsi condensée n’est pas transcriptionnelle, à savoir qu’elle ne sert pas à la création d’ARNm et de protéines. C’est l’ADN située entre les nucléosomes qui sert à la transcription en ARNm pour créer des protéines. Par contre, lors de la division cellulaire, l’ensemble de l’ADN y compris la “non transcriptionnelle” est copiée à l’identique.
Nucléofilament
C’est le niveau de compaction suivant de la chromatine qui voit la suite d’ADN poncturée de nucléosomes s’enrouler elle-même sous forme de solénoïdes. On a un nucléosome toutes les 200 paires d’ADN ce qui veut dire qu’ils sont les uns contre les autres. Ces nucléofilaments font environ 30 nm de diamètre. Eux-mêmes sont ensuite compactés au sein des chromosomes mais leur compactage n’est pas le même pendant le cycle de vie de la cellule. La forme la plus compactée se manifeste lors du processus de division cellulaire au moment de la prophase. Mais la réplication de l’ADN dite semi-conservative (ou la moitié du brin est conservée et chaque moitié complétée par un réplicat en négatif) a lieu pendant l’interphase, soit la vie “normale” de la cellule. En temps normal pendant l’interphase, les nucléofilaments s’étalent de manière libre au sein du noyau des cellules.
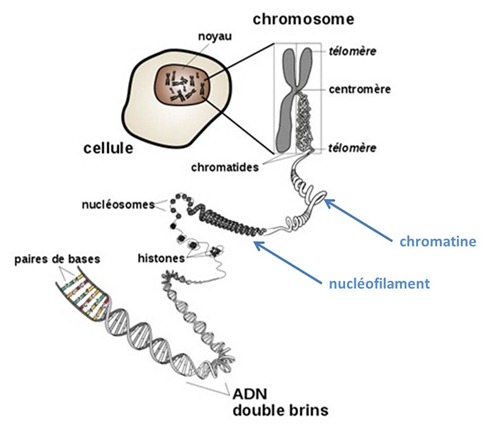
Chromatine
C’est la forme sous laquelle se présente l’ADN dans le noyau des cellules et les chromosomes. Cette substance de base des chromosomes associe un brin d’ADN, de l’ARN et des protéines. Dans la chromatine, on distingue l’euchromatine qui contient la partie active de l’ADN, utilisée lors de la transcription en ARNm et en protéines et se situe généralement entre les nucléosomes. Elle représente 10% de l’ADN chez l’homme et comprend de 23000 à 25000 gènes (27% du code), de l’ADN non codant répété (50%), de l’ADN non codant non répété (4%), de l’ADN codant dupliqué (7%) et des séquences indéterminées qui sont inclassifiables à ce stade des connaissances (5%). Il y a ensuite l’hétérochromatine qui est de l’ADN condensé sous forme de fibres de 20nm à 30nm de diamètre mais qui ne sert pas à la transcription de l’ADN en ARNm et en protéines.
Chromosomes
Ce sont les longues molécules de chromatine intégrant de l’ADN. Leur forme évolue lors du processus de reproduction des cellules comme indiqué ci-dessus. En temps normal, en fait, pendant la période dite G1 de l’interphase, un chromosome humain est un fil qui s’étend de part et d’autre d’un centromère, une longue chaine d’ADN, de plusieurs milliers de paires de bases d’ADN chez l’humain, et dont la chromatine utilise une histone particulière. Le centromère fait le lien entre les chromatines d’ADN qui se dupliquent lors de la division cellulaire.
La forme en X bien connue n’a lieu que lors de la reproduction des cellules dans une période particulière de cette division qui s’appelle la métaphase et qui dure environ 10 minutes. L’une des raisons pour lesquelles c’est la manière dont on représente le plus fréquemment les chromosomes comme ci-dessous est que c’est la plus facile à photographier ! Les autres ? Je ne sais pas…
A l’extrémité des chromosomes se trouvent les télomères (découverts en 1984 par les américains Blackburn, Greider et Szostak), des séquences non codantes d’ADN qui sont répétées plusieurs fois. Ces télomères se raccourcissent à chaque division cellulaire pour la plupart des cellules humaines. Elles sont ainsi un marqueur de l’âge de l’organisme vivant. Elles comportent jusqu’à 12000 paires de base d’ADN chez le nouveau né et tombent à moins de 6000 paires pour l’octogénaire en bonne santé. Mais les globules blancs ainsi que les cellules cancéreuses quoi se dupliquent fréquemment et rapidement bénéficient d’une enzyme particulière qui conservent leurs télomères. Des recherches visant à limiter le phénomène de raccourcissement pour les cellules saines pourraient aboutir à la création d’élixirs de jouvence, à base de télomérase ! Question taille, le premier chromosome humain mesure 245 millions de bases x 0,34 nm (nanomètre), soient environ 8 cm, une fois déroulé ! En pratique, un chromosome humain fait 1400nm (1,4 micron) de diamètre pour environ 10 microns de long.
Génome
Décrit le patrimoine générique d’une cellule qui se matérialise sous la forme de l’ensemble des gènes que l’on trouve à la fois dans le noyau de la cellule avec ses chromosomes mais aussi, pour une part négligeable, dans ses mitochondries.
Nucléole
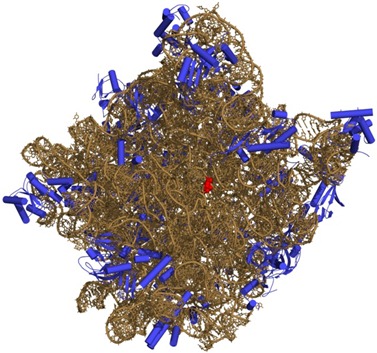
Partie du noyau des cellules où se produit la transcription des ARN ribosomiques (ARNr, issues de la transposition de l’ADN), qui constituent avec des protéines, les deux sous-unités des ribosomes (exemple ci-dessous), les molécules complexes qui servent elles-mêmes à synthétiser les protéines à partir du code compris dans l’ARNm.
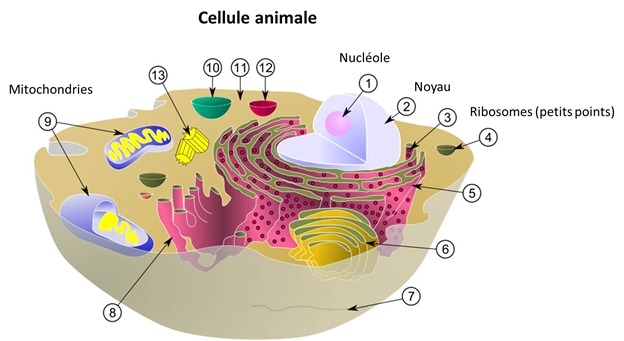
Noyau de la cellule
Il contient les 23 chromosomes avec l’essentiel du génome humain, qui sont sous forme de paires de chromosomes entre la phase S1 de l’interphase et la métaphase. C’est au sein du noyau que l’ADN est à la fois dupliquée pendant la division cellulaire et aussi, qu’il donne lieu à la création des différentes formes d’ARN, et notamment l’ARNm qui contient un double du code des gènes et servira ensuite dans le cytoplasme (le reste de la cellule) à générer les protéines, aidé par les ribosomes et les ARN de transfert.
Mitochondries
Composantes des cellules, elles contiennent aussi un petit bout du code ADN des cellules avec 16 kilobases organisées dans un génome circulaire et 37 gènes qui “codent” 13 protéines, 22 ARN de transfert et 2 ARN ribosomiques. Le décodage de cette partie du génome permet de vérifier les filiations mère-enfants et de dater les lignées car chez l’homme, ces gènes sont transmis uniquement par la mère.
Cellule dites “Eucaryotes”
Que l’on trouve dans les espèces animales et végétales qui comprennent entre autres un noyau et des mitochondries. Et toute une artillerie de domaines spécialisés. Ce sont de véritables usines chimiques qui transforment l’énergie, dans un sens pour l’emmagasiner et dans l’autre pour la restituer de manière chimique ou mécanique, comme dans les muscles.
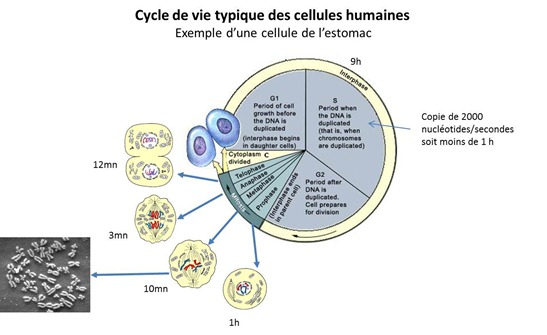
Dimension temps
Les cellules humaines ne suivent pas toutes le même métabolisme. Certaines se reproduisent par division cellulaires mais d’autres non, comme les globules rouges, les cellules musculaires du cœur ainsi que les nerfs. La durée de vie des cellules qui se reproduisent va de quelques journées (dans la peau ou le système digestif) à plusieurs mois voire années (dans le pancréas ou les os). On verra dans un prochain article à quel stade de développement des cellules humaines on prélève leur ADN pour leur séquençage.
Comparaison de tailles
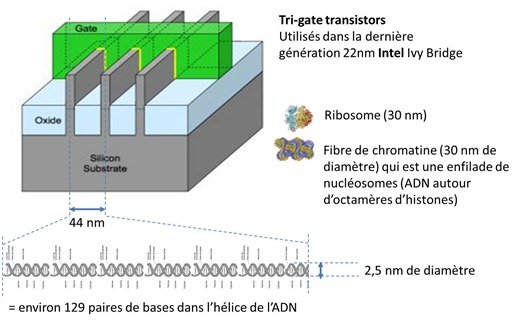
Chaque paire de bases de l’ADN est espacée de 0,34 nm et l’hélice de l’ADN fait 2 nm de diamètre. Un tour complet de l’hélice d’ADN se fait en 10 paires de bases. Comment cela se compare-t-il avec les microprocesseurs les plus récents en termes d’intégration ?
Si on prend comme référence la dernière génération de processeurs 22nm de la série de processeurs Core “Ivy Bridge” d’Intel, chaque transistor est espacé de 44 nm sur son substrat en silicium, le niveau d’intégration “22nm” correspondant au demi-espacement entre transistors. En faisant un petit calcul simple, on constate que cet espacement correspond à l’enfilement linéaire de 129 paires de bases d’ADN.
Mais l’ADN s’enroule de manière complexe autour d’histones, elle-même compactées dans des fibres de chromatine ce qui fait que la densité du code génétique est encore plus forte que dans cette vue linéaire. Ainsi l’espace entre transistors dans ces processeurs (44nm) est-il voisin du diamètre (33nm) des fibres de chromatine.
En termes de condensation d’information, l’ADN est donc bien plus dense que ces microprocesseurs ou les mémoires qui utilisent des technologies similaires à base de silicium. Ainsi, le petit bout de fibre de chromatine dans le schéma ci-dessous qui s’insèrerait entre deux transistors 22nm comprend-il 12 nucléosomes avec 140 paires de bases d’ADN chacun, soit en tout 1680 paires. Chaque paire de base est en équivalent informatique un code à 2 bits (puisqu’il y a quatre possibilités), ce qui donne 3 Kbits en tout.
A vrai dire, c’est avec la densité d’un disque dur qu’il faudrait faire cette comparaison plutôt qu’avec un processeur. Faisons-là donc. Dans les disques durs actuels de 2 To de 3,5 pouces utilisant la technologie de stockage perpendiculaire magnétique (PMR) co-inventée par le prix Nobel français Albert Fert, la distance entre chaque bit sur les plateaux magnétiques est d’environ 32 nm – sans rapport avec les technologies d’intégration de silicium en 32 nm.
Elle va descendre à court terme à 25nm et en dessous grâce à la technologie Heat-Assisted Magnetic Recording (HAMR) qui s’appuie sur l’usage d’un laser. Mais cette technologie pourrait en théorie faire descendre la distance entre bits à 3,6 nm, soit l’ordre de grandeur de l’épaisseur d’un brin d’ADN. Sachant qu’en parallèle, l’espace entre transistors dans les circuits intégrés pourrait descendre à 20 nm (en technologie 10 nm).
Le défi du séquençage
Reste à décoder tout cela… ce n’était que l’apéritif pour comprendre la suite des événements. Alors, qu’est-ce que le séquençage du génome humain ?
Il s’agit de décoder l’ADN de nos chromosomes, soit plusieurs milliards de paires de bases notées A, G, C et T sachant on l’a vu qu’elles sont intégrées dans des chromosomes qui ne se présentent pas linéairement mais en fibres elles-mêmes constituées de nucléosomes. Et puis, ces chromosomes sont dans le noyau de nos cellules. Il faut les en extraire !
Au départ, l’objectif était de se concentrer sur le décodage des gènes, la petite partie de l’ADN qui code la création des protéines, le reste de l’ADN ayant une fonction autre, essentiellement de “support” au sein de la chromatine et des chromosomes. Mais le séquençage du génome humain est tout de même allé au delà et a couvert l’ensemble de l’ADN de nos chromosomes. La raison est que les séquences non codantes qui sont parfois redondantes et répétitives présentent aussi un grand intérêt scientifique.
Première utilité, identifier les séquences dites régulatrices qui influencent l’expression des gènes et notamment dans les maladies d’origine génétiques. On a encore beaucoup à apprendre et découvrir de ce côté-là. Ce d’autant plus que les séquences régulatrices d’un gène peuvent se trouver n’importe où dans le génome et pas simplement à proximité des gènes en question.
Autre utilité : appréhender l’origine de mutations dans les espèces et retracer tout simplement l’histoire de la vie sur terre. Cela sera d’une plus grande utilité pratique lorsque l’on se sera attaqué à un séquençage du génome de nombreuses espèces animales et végétales car aujourd’hui seules quelques dizaines ont été traitées et plutôt pour des bactéries et autres organismes monocellulaires. Mais le séquençage du génome du poulet, d’une espèce de cochon domestique et du bœuf ont déjà été réalisés dans les années 2000. Un séquençage qui permet de s’attaquer aux dizaines de milliers d’agents pathogènes de ces animaux domestiqués afin de mieux traiter les épidémies les frappant. Avec des conséquences macro-économiques et sociétales majeures. Mais la comparaison de l’ADN des espèces animales avec l’ADN humaine permet aussi d’en savoir plus sur les séquences régulatrices.
En guise de teasing pour la suite, pour faire simple, le procédé du séquençage consiste à d’abord à débarrasser l’ADN des protéines qui l’entourent par traitement chimique puis à la découper en morceaux. Chaque morceau est à son tour découpé en morceaux de taille variable, à une base près. Et un procédé à base de capteurs de fluorescence permet d’identifier le nombre de morceaux de chaque taille et la nature de la base à son bout. On en déduit un grand nombre de séquences d’ADN redondantes qui se recouvrent. C’est alors par logiciel que l’on rassemble toutes ces séquences pour reconstituer pas à pas le génome humain. Le volume de données est raisonnable (3 milliards de bases au plus), mais c’est ce traitement de recombinaison des séquences qui est très lourd. Et ensuite, son exploitation.
Ça, c’est l’explication rapide. On en détaillera le fonctionnement dans l’article suivant de cette série. Et on examinera les machines qui réalisent ce séquençage du génome très rapidement en s’appuyant sur des techniques de séquençage massivement parallèles.
![]()
![]()
![]()
Reçevez par email les alertes de parution de nouveaux articles :
![]()
![]()
![]()
































 Articles
Articles
Superbe idée de série d’articles! j’adore quand on relie deux grands domaines scientifiques, c’est comme cela que se créent les grandes vocations.
L’idée de comparer la densité à celle d’un disque dur est originale. Tu devrais t’intéresser à l’épigénétique, c’est un bon sujet à la périphérie de celui que tu abordes.
Sinon, attention, le C de la cytosine est devenu un C de copyright
Bonjour Hervé,
Merci pour l’encouragement car c’est un domaine assez particulier à appréhender. On est vite noyé sous les concepts. La biologie moléculaire est d’une richesse incroyable !
L’épigénétique ? Je vais voir cela dans les phases suivantes, peut-être au niveau des applications du séquençage.
Pour le (c), corrigé : c’était une fonction à désactiver dans un plug-in !
Excellente série à lire cet été: “Les technologies du séquençage du génome humain – 1” de @olivez sur http://t.co/GNoQrqG2
Sur ce sujet, il va forcement falloir passer par les technologies de séquençage de différentes génération.
Ce n’est pas vraiment évident à comprendre, surtout les appareils de secondes générations qui font du reséquençage.
Si vous avez besoin, j’ai un document sur le sujet que j’avais préparé pour un laboratoire d’analyse. Je ne suis pas sur qu’il soit forcement super complet, mais ça fait une bonne base pour recherche de l’information.
J’avais contacter à l’époque les différents producteurs. N’hésitez pas à me contacter si vous en avez besoin. Je regarderais si je l’ai encore.
Bonjour, oui je suis preneur de toute littérature pour débroussailler le sujet ! Merci !
excellent et beau travail
(et je viens enfin de comprendre le rapport entre ADN et Chromosome, dont le lien a été pour ainsi dire flou jusqu’ici !!!). La chromatine nous a sauvé !
Et bam, je viens de tenter de me refaire tout le programme de Terminale S en 15 min de lecture. Bon rafraichissement et des compléments intéressants.
En revanche cette lecture ne me suffit pas à ancrer les informations assez profondément quand bien même les sillons existent déjà quelques parts dans mon cerveau. Pour bien faire il faudrait en passer par la même pratique que vous: lire, assimiler, enrichir et reformuler.
Vous êtes vous toujours plié à ce genre d’exercice ou avez vous commencé avec votre Blog ?
Je fais cela assez régulièrement, mais habituellement sur les sujets qui sont dans mon coeur de métier (médias numériques). Ici, c’est une digression d’été qui m’est venue car je souhaitais comprendre comment on séquençait le génome. La variété des technologies mises en oeuvre est assez fascinante.
“Les technologies de séquençage du génome humain – 1” de @olivez sur http://t.co/GfxSC83X
Allez un peu de courage et on profite de ce passionnant résumé didactique d’@olivez sur le séquençage du génome humain: http://t.co/Pw0uyfi8
Pour aller plus loin sur la comparaison processeur/stockage: l’ADN comme support de stockage: http://hms.harvard.edu/content/writing-book-dna
Merci ! C’est en fait la chute de la 5ième partie de la série, ici : http://www.oezratty.net/wordpress/2012/technologies-sequencage-genome-humain-5/.
Oups… Avais pas terminé la série! J’y vais de ce pas
merci beaucoup à vous pour ces articles intéressants pour moi et très détaillés: les technologies de séquencages du génome humain
mercii 🙂
passionnant ! “Les technologies de séquençage du génome humain – 1” de @olivez sur https://t.co/SATPgC0eaV