Après avoir couvert la méthode de Sanger, le séquençage par terminateur réversible et le pyroséquençage, poursuivons notre découverte des technologies de séquençage de l’ADN démarrée ici. Vous verrez que la variété des techniques mises en œuvre est très grande et que la créativité dans ce domaine est assez fascinante.
Ces technologies impliquent une polymérisation de l’ADN sous une forme ou une autre pour identifier les bases ajoutées, ou bien pas de polymérisation du tout et une détection directe des bases de l’ADN par procédé optoélectronique ou électronique. Côté captation de l’information, tout y passe : des capteurs CCD complétés ou pas de lasers, de la microscopie électronique, et de la mesure électronique de conductance ou de pH.
Un grand nombre de ces technologies n’en sont qu’à l’état de prototype et ne sont donc pas encore commercialisées. Les startups en question annoncent toutes une commercialisation entre fin 2012 et 2013. Et les délais dans ce domaine sont aussi si ce n’est plus élastiques que dans le logiciel car il faut maitriser simultanément des questions de chimie, de fluidique, d’électronique et de calcul, sans compter les questions d’industrialisation du matériel !
Séquençage par ligation (SOLID technology de Life Technologies)
La technologie SOLID est issue de la société Agencourt Personal Genomics et a été acquise par Applied Biosystem en 2006, elle-même fusionnée ensuite avec Life Technologies. SOLID veut dire ”Sequencing by Oligonucleotide Ligation and Detection”. Le processus démarre un peu comme pour le pyroséquençage mode 454 Roche, à savoir qu’une bibliothèque de brins d’ADN est associée à une microbille magnétique qui fait ici 1 micron d’épaisseur via un adaptateur (petit brin d’ADN) et que chaque brin est multiplié par emPCR. Le résultat est fixé sur une plaque de verre et non dans des micro-cuves. Chaque brin est associé à une amorce pour pouvoir initialiser sa réplication.
C’est alors qu’a lieu une série d’opérations chimiques assez ingénieuses pour répliquer ces brins d’ADN et détecter au fur et à mesure les bases qui sont ajoutées. Il s’appuie sur la ligation, un processus habituellement utilisé pour la réparation (automatique) de l’ADN des cellules, notamment lors de sa réplication (naturelle). Une protéine complexe, la ligase, est capable de raccommoder des morceaux d’ADN les uns aux autres.
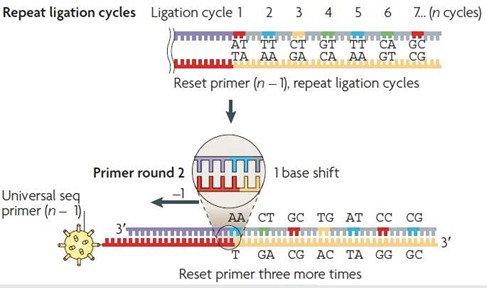
Le processus consiste à ajouter par ligase et de manière répétitive, des blocs de 8 bases avec une paire de bases qui correspondront aux deux suivantes à synthétiser dans chaque brin d’ADN à séquencer (parmi 16 possibles, 4×4), précédée de trois bases degenerate et suivie de trois bases universelles et d’un marqueur au fluor qui seront éliminées. Des bases degenerate et universelles ? Il s’agit de bases qui sont capables de s’apparier avec n’importe laquelle des quatre bases de l’ADN. Comme la deoxyinosine, une base G sans son amine (NH2).
Les 16 paires de bases différentes que l’on va assembler à l’ADN à séquencer sont marquées par fluorescence. Mais avec 16 couleurs différentes ? Non, ce serait trop simple. On utilise seulement quatre couleurs différentes car visiblement il est délicat d’en utiliser plus en biochimie. Ce qui veut dire que la détection de la couleur donne une information partielle sur la nature de la paire de bases détectée. L’astuce consiste à ajouter donc pas à pas des séries de paires de bases détectées par fluorescence suivies de 3 bases universelles, d’enlever le tout et de recommencer l’opération quatre fois en la décalant d’une base, grâce à des amorces de taille différente (de 1 à 4 bases en moins que la première). Cela permet par recoupement d’identifier les bases de l’ADN et qui plus est, de les mesurer chacune deux fois. Ce qui réduit le niveau des erreurs de lecture à 1 pour 1000, soit dix fois mieux que dans le pyroséquençage. C’est l’aspect le plus différentiant de cette technologique.
Tout ceci est un peu compliqué à piger. Il faut en tout cas retenir que ce système est capable de lire un milliard de bases par batch ce qui est un niveau très concurrentiel à ce jour.
Et la machine ? La voici, et elle coute environ $500K. Petit détail, je n’ai pas pu savoir dans la littérature correspondante comment fonctionnait le capteur d’image de la plaque de verre utilisée pour le séquençage ni quoi que ce soit sur sa taille. On va supposer qu’il s’agit d’un capteur d’image classique ou bien voisin de celui qui est exploité dans la machine GS FLX Titanium XL+ de Roche. Par contre, on sait que le bas de la machine comprend un PC sous Linux avec 13 Teraoctets de stockage pour enregistrer toutes les images générées par la lecture du séquençage base par base et ensuite reconstruire l’ADN par calcul. Les données brutes “image” d’un séquençage de ce type prennent en effet beaucoup de place !
Séquençage par semiconducteur (Ion Torrent / Life Technologies)
La Proton de Ion Torrent est un séquenceur qui ne s’appuie pas sur la détection de fluorescence de nucléotides ou de leurs résidus de polymérisation par un capteur optique CCD. Il utilise un capteur CMOS qui détecte non pas des photons mais des ions, plus précisément les ions H+ qui sont dégagés lors de la polymérisation de l’ADN. Un ion H+ est en fait un simple proton qui gambade sans électron !
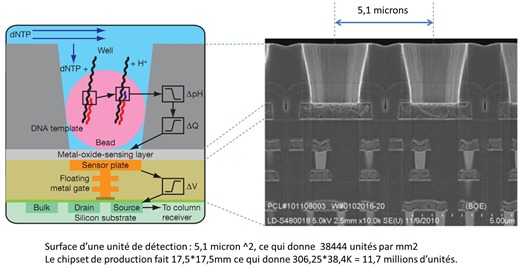
Ce capteur est associé à des micro-puits comme pour le séquençage 454. Le capteur CMOS mesure le pH (l’acidité) dans chacune des cavités, ce qui indique la présence d’une ou de plusieurs bonnes bases qui ont été intégrées dans l’ADN en cours d’analyse. Chaque cavité permet de séquencer un brin d’ADN d’environ 200 bases. Le processus est sinon voisin du pyroséquençage avec ajout des bases les unes après les autres pour “voir” laquelle “prend” dans chaque cavité. Puis rinçage par microfluidique et passage à la base suivante à tester.
La première version expérimentale du chipset CMOS de Ion Torrent comprenait 25 électrodes de 300×500 microns sur une surface de 5,3 mm2. Sont arrivés ensuite les chipsets 314, 316 et 318, le dernier comprenant 11 millions d’unités de détection. Il sont intégrés dans le séquenceur Ion PGM Sequencer. Le chipset 318 fait 17,5mm2 de côté avec des unités de détection de 5,1 microns de côté. Ce qui donne 11,7 millions d’unités de détection. Elles sont produites sur des wafers de 8 pouces comportant 200 chipsets. Les chipsets sont combinés dans les machines à de la microfluidique pour l’alimentation en ADN et en agents de séquençage.
Ce niveau d’intégration est donc de deux ordres de grandeur plus grands qu’avec les machines de pyroséquençage 454 de Roche. On passe en effet de 54 microns à 5,1 microns pour la largeur des unités de détection de polymérisation d’ADN. L’intégration est donc de (54/5,1)^2 plus grande, soit exactement 112 fois. Mais Ion annonce être capable à assez court terme de fabriquer des capteur CMOS avec un milliard d’unités de détection. Par contre, les brins d’ADN séquençables sont un peu plus courts et font un maximum de 200 bases. Les batches de séquençage durent 2 à 4 heures.
Mais ce n’est pas tout. Les séquenceurs Ion Proton sont dotés d’une nouvelle génération de chipsets, le Proton I et le Proton II qui comprennent respectivement 165 et 660 millions d’unités de détection. En un seul batch de deux heures, ce dernier peut séquencer des brins d’ADN de 100 bases en moyenne. Ce qui donne 66 Giga bases. Comme le génome humain en comporte 3, cela donne un taux de couverture de plus de 30x, ce qui est excellent mais qui sous-entend un taux d’erreurs significatif. Il est notamment lié au nombre relativement restreint de bases que les brins analysés peuvent comporter (100 à 200). Plus il est faible, plus il est difficile de reconstituer le puzzle de l’ADN par traitement numérique.
La société Ion Torrent avait été fondée en 2007 par Jonathan Rothberg, l’un des créateurs de 454 Life Science Corp qui l’avait revendu à Roche pour $140m. Ion Torrent a été acquise par la société californienne Life Technologies pour $725m en 2010. La même qui commercialise la technologie SOLID, après une fusion avec Applied Biosystems opérée en 2008. Ce groupe fait un CA annuel d’environ $4B (4 milliards de dollars) et emploie plus de 10000 personnes. On est loin de la startup !
SMRT (Single Molecule Real-Time Analysis de Pacific Bioscience, $250m levés)
Cette technologie utilise le marquage par fluorescence couleur des nucléotides ajoutées aux brins d’ADN transcrits par polymérase. Leur ajout est détecté en véritable temps réel au fur et à mesure de leur ajout au brin d’ADN à séquencer.
Son bénéfice principal est de permettre de lire d’une traite des séquences allant jusqu’à 3000 bases, pour 100 à 1000 pour les technologies que nous avons vues jusqu’à présent. Cela contribue à diminuer le nombre d’erreurs et à réduire le niveau de taux de couverture (# de bases à détecter par redondance / nombre de bases de l’ADN à séquencer). Un batch dure moins d’une journée.
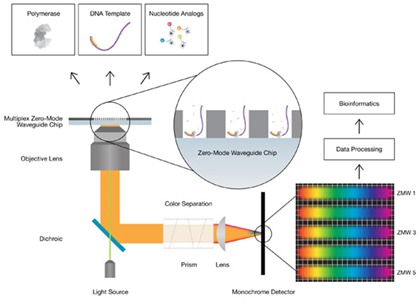
La “PacBio RS” (machine ci-dessus) utilise des chipsets nanofluidiques qui contiennent des petits trous de quelques dizaines de nanomètres d’épaisseur au fond desquels est attaché une molécule d’ADN polymérase qui sert à gérer une transcription contrôlée de brin d’ADN. C’est l’ADN qui va se déplacer base par base sur cette ADN polymérase et non le contraire.
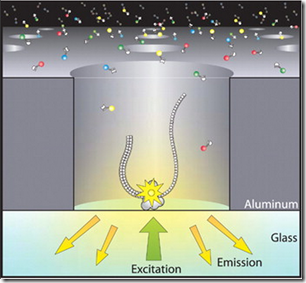
Des nucléotides marqués par fluorescence sont intégrés dans l’ADN en cours de transcription. Les cavités du chipset appelées “zero mode waveguides” vont capter les quelques photons émis par la fluorescence de la nucléotide intégrée dans l’ADN, qui est activée par rayon laser, et la router via des fibres optiques vers un capteur CCD (schéma ci-dessous).
Les chipsets SMRT contiendraient actuellement 150000 cavités. C’est peu au regard des autres systèmes de séquençage massivement parallèles. Mais celui-ci fonctionne réellement en temps réel : il ne faut pas attendre 2 minutes entre chaque ajout de base pour faire le ménage avec des solvants. Ici, les bases sont incorporées les unes après les autres. Entre chaque incorporation s’écoulent seulement quelques millisecondes.
Séquençage à l’aide de nanopores (GeniaChip, BioNano Genomics, NABsys, NobleGen, Oxford Nanopore Technologies, Stratos Genomics)
Cette technologie est encore plus ébouriffante que les précédentes. Elle consiste à faire passer des brins simples ou doubles d’ADN au travers de trous, des nano-pores, et à lire les bases qui les traversent une par une, ou bien des sondes ou marqueurs associés. Nuance cependant : les séquenceurs utilisant ces technologies ne semblent pas encore au point et relèvent encore de l’effet d’annonce. Les nanopores sont réalisés soit à base de protéines soit sur circuit intégré à base de silicium. Soit les deux.
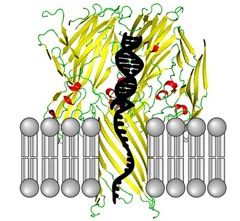
Ce genre de technologie est notamment proposé par GeniaChip (Mountain View, créée en 2009) qui exploite des brins simples d’ADN (ssDNA ou single stranded DNA). Lorsque ces brins traversent des pores de 2 nanomètres de diamètre qui sont en fait des sites moléculaires actifs (ci-dessous), ils modifient le courant électrique qui les traverse, et cela tombe bien, ce courant dépend de la base d’ADN. Les nano-pores sont faites de protéines lipidiques installées sur un substrat silicium. La technologie présente plein d’avantages : on n’a pas besoin d’ajouter tout un tas de composants chimiques pour entretenir la réaction, elle est très rapide et il n’y a pas d’amplification d’ADN génératrice d’erreurs. Elle pourrait permettre à terme de séquencer le génome humain en moins d’un quart d’heure… hors traitements informatiques qui pourraient alors représenter le gros du temps du séquençage.
Avec celle de Ion Torrent, c’est la seconde technologique de séquençage “non optique” examinée dans ces lignes. Le processus est très rapide puisqu’une base peut être lue toutes les 10 nanosecondes ! Comme la mesure est électrique, l’électronique peut suivre, ce qui ne sera pas le cas avec une mesure optique et un capteur CMOS ou CCD. Seul problème de taille à régler : la mesure est entachée de nombreuses erreurs qu’il faut ensuite corriger. Sans compter la mécanique, en général électrique, pour faire passer le brin d’ADN au travers des nano-chats d’aiguille ! C’est pourquoi d’autres sociétés cherchent à soit ralentir le processus soit à augmenter la taille des éléments mesurés au travers des nanopores.
Chez NABsys (Providence, Rhode Island, $21m levés), on utilise ainsi une technique de “positional sequencing” qui hybride de longs fragments d’ADN simples de plus de 100K bases à différentes positions avec une sonde qui combine six bases d’ADN et une molécule complexe pour sa détection. L’ensemble est complété avec le reste de l’autre brin et des protéines d’assemblage. Il passe ensuite au travers d’un nanopore qui détermine la position des sondes par conductivité électrique et au rythme de un million de bases par seconde. Ces positions sont entachées d’erreurs qui sont corrigées ensuite par calcul et en utilisant la redondance du système.
Il faut réaliser plusieurs fois cette opération sur le même brin d’ADN et avec des sondes différentes de 6 bases permettant de bien couvrir l’ADN. Il en faudrait 4096 en théorie pour couvrir toutes les combinaisons de 6 bases d’affilée (4 puissance 6). Les capteurs qui comportent des dizaines de milliers de nanopores par cm2 sont conçus avec des technologies de fabrication de semi-conducteurs traditionnelles et en mode “fabless” (sans usine) comme il se doit. Les chipsets peuvent même fonctionner en parallèle, pour créer des systèmes de séquençage de masse extrêmement rapides. La technologie est actuellement au stade de l’industrialisation. Traduction : pas encore commercialisée.
Il y a aussi l’américain PathoGenetix qui propose une technique de séquençage qui rappelle celle de Nabsys mais utilise des tags de 3 bases en lieu et place de 6 bases, qui sont détectés ensuite par passage des brins d‘ADN greffés dans un système de microfluidique. Cela permet de faire passer 150 millions de bases par secondes. Le système qui a été financé à hauteur de $50 par le DHS (Department of Homeland Security), un record de financement public dans le secteur, semble viser non pas le séquençage complet de génome mais la détection de variations pour l’identification de personnes.
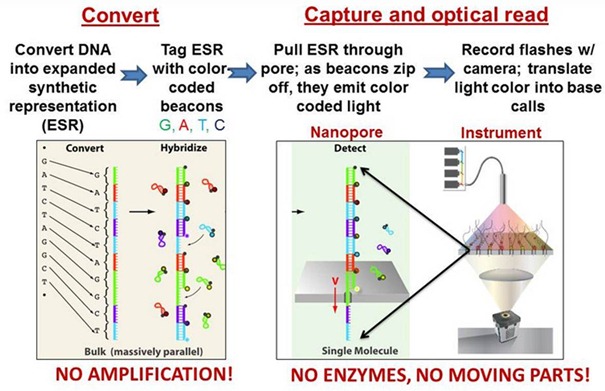
NobleGen BioSciences est une startup américaine créée par un chercheur et ayant reçu $12,5m de financements publics via la NSF et différents concours.Elle utilise aussi des nanopores mais avec encore un autre procédé. L’ADN à séquencer est d’abord transformé en une molécule synthétique où chaque base d’origine est convertie en une séquence de nucléotides unique qui est appariée à une molécule complémentaire dotée d’un fluorophore de couleur unique et d’un complément (beacon). C’est une sorte d’inverse de la compression en numérique qui est ici utile pour améliorer la précision de la mesure. Cela donne une molécule à double-brin ressemblant à l’ADN qui est poussée dans le nanopore. Parce que les nanopores sont trop étroits, les marqueurs fluorescents sont éliminés, ce qui génère une lumière qui est captée optiquement et via une excitation par un laser. Le processus permet en pratique de décoder 96 génomes différents en 17 heures.
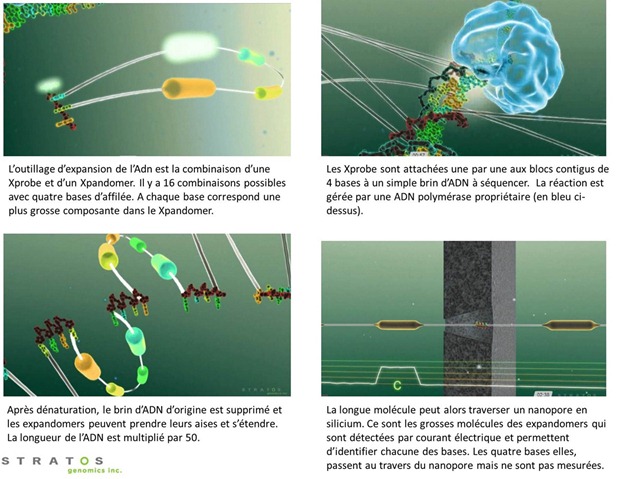
Stratos Genomics (Seattle, $7m levés) utilise une technologie différente mais avec la même finalité : réduire les erreurs de lecture au travers des nanopores en rallongeant artificiellement la taille des bouts d’ADN à analyser. Ici, d’un facteur 50 bien plus grand qu’avec la technologie de NobleGen. Elle intercale toutes les quatre bases d’un brin d’ADN double branche une molécule appelée ‘xpandomer’ via un processus voisin de la réplication qui utilise une polymérase spécifique. L’autre brin de l’ADN est enlevé par dénaturation, plus précisément, par chauffage. Les ‘expandomer’ s’étendent comme leur nom l’indique pour rallonger l’ADN à séquencer. Tout ceci est expliqué dans un brevet si vous avez le courage de vous le farcir. Ca a l’air bien astucieux mais aucune caractéristique précise du système n’est pour l’instant indiquée par le concepteur au-delà de dire qu’il peut séquencer 10 millions de bases par seconde. Le système n’est bien évidemment pas plus disponible que les autres dans cette catégorie “nanopores”.
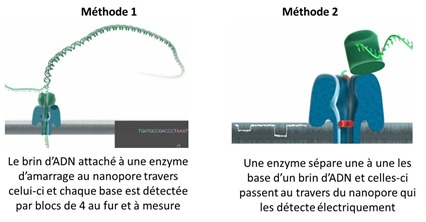
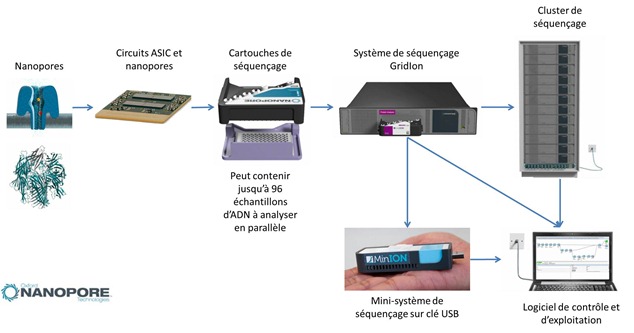
Oxford Nanopore Technologies (Oxford UK et USA, $120m levés, avec investissement d’Illumina, création en 2005, plus de 300 brevets déposés) et son système GridION permet de séquencer des molécules d’ADN à simple brin selon deux méthodes différentes. Dans les deux cas, les nanopores utilisés sont à base de protéines complexes déposées sur une membrane synthétique en polymère. Le tout est posé sur un chipset produit en ASIC (circuit intégré conçu pour une seule application). L’ensemble est dans une cartouche de consommable.
Dans la première méthode, un ADN double-branche est accroché à une enzyme qui va s’amarrer au nanopore et déclencher le passage de l’un des brins de l’ADN dans celui-ci puis le second brin. Les bases qui traversent le nanopore sont détectées électriquement comme avec le GeniaChip, mais semble-t-il, par blocs de 4 ce qui réduit les erreurs. Quand le premier brin a été analysé, l’autre brin l’est également (animation), et dans l’autre sens, ce qui créé une information redondante limitant les erreurs de séquençage. Dans une seconde méthode, une exonucléase (enzyme qui coupe l’ADN un nucléotide à la fois) est attachée à la fois à l’ADN et au nanopore. Elle déplume l’ADN à séquencer base par base. Chaque base enlevée passe au travers d’une cyclodextrine, une molécule intégrée dans le nanopore, et est détectée électriquement. Comme elles sont bien séparées les unes des autres, les risques d’erreurs sont limités.
La machine serait commercialisée à $30K. au format 2U, elle est conçue pour fonctionner de manière parallèle avec plusieurs unités contrôlées de manière unique dans des racks, comme dans un datacenter. Les données pouvant être uploadées à distance et contrôlées dans une application fonctionnant dans un navigateur. Comme les résultats sont récupérés en temps réel, on peut arrêter le séquençage lorsque l’on a obtenu assez de données, ce qui fait gagner du temps. Ce genre d’outil est clairement destiné à une exploitation industrielle à grande échelle et pas que pour faire de la recherche. A l’autre extrémité, la société prévoit de sortir un mini-séquenceur qui tiendra dans une simple clé USB et qui sera commercialisé à $900. Impressionnant ! Cette société a obtenu l’un des plus gros financements en capital risque de toutes celles que je suis en train de couvrir ici. La technologie devrait être disponible d’ici début 2013.
Enfin, chez BioNano Genomics (Philadelphie, $33m levés, créé en 2003), on a inventé un système de micro-canaux permet de débobiner progressivement un brin d’ADN et de le faire passer dans un nanotube en longueur. Le brin est marqué de diverses manières par fluorescence et un détecteur optique permet d’identifier les différentes parties de ce brin. Avec cette technologie, les brins d’ADN peuvent comporter jusqu’à un million de paires de bases. Un système électrique fait avancer l’ADN pas à pas dans les nano-tubes et un capteur CCD (le nanoAnalyzerCCD) détecte les marqueurs qui passent. Ces marqueurs permettent d’identifier des gènes ou des modifications de zones connues. Mais pas les séquences de bases à proprement parler. Les marqueurs permettent de visualiser la structure d’ensemble de l’ADN pour en faire une sorte de cartographie. Un génome humain va pouvoir être découpé (avec des enzymes de restriction bien choisies) en quelques milliers de brins d’un million de paires de bases qui vont être analysés en parallèles en moins de 10 minutes. Ce qui semble très prometteur. La technologie est cependant prometteuse : elle pourrait être couplée à un système de détection de bases et pourquoi pas à celui de GeniaChip. D’où des travaux réalisés avec une autre société, Complete Genomics, pour réaliser le séquençage des brins qui ont été alignés dans le composant.
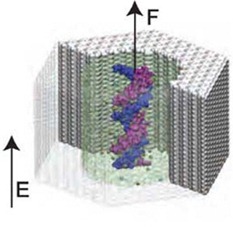
On pourrait aussi citer IBM dont les laboratoires de recherche planchent sur la question de l’optimisation du séquençage par nanopores avec ses DNA transistors (ci-dessous). Leur technique s’appuie sur des nanopores en semi-conducteurs qui utilisent des couches alternées de métal et de diélectrique (isolant) permettant par le courant électrique qui traverse les premiers de contrôler l’avancement de l’ADN dans le nanopore, et ensuite, de mesure la présence des quatre bases. Le projet avait été présenté en 2009 et depuis, plus de nouvelles ! Et on ne l’a pas vu réapparaitre sous la forme de technologie employée sous licence chez d’autres fabricants. Cela veut dire que cela ne fonctionne peut-être pas de manière satisfaisante !
Voilà pour les nanopores. Difficile de départager toutes ces technologies. Certaines produisent moins d’erreurs que les autres en lecture, mais celles qui en créent le plus ont toutes des dispositifs de redondance et de calcul pour les réduire à la portion congrue dans les résultats finis.
Analyse par microscopie électronique (Electron Optica, Reveo et ZS Genetics)
Voici un autre groupe de technologies qui consiste à utiliser un microscope électronique pour détecter les bases d’ADN. Pourquoi pas si l’engin n’est pas trop lourd ! Il y a un hic dans l’histoire : aucune des sociétés de ce créneau n’arrive à mettre au point sa technologie, à lever suffisamment de fonds et à être compétitive par rapport aux autres technologies de seconde et troisième génération.
Quelques sociétés identifiées dans ce secteur : Electron Optica dont le matériel est fabriqué par la société tchèque Delong Instruments, Reveo et son Omni Molecular Recognizer Application (OmniMoRA) qui utilise des sondes d’excitation des bases pour les rendre visibles au microscope électronique, et ZS genetics qui incorpore des labels ‘ZSG’ rendant les bases détectables au microscope électronique. Dans ces trois cas, il existe très peu de littérature ou de description du matériel de séquençage.
On pourrait par extension citer également Lightspeed Genomics (anciennement Solametrix, Santa Clara, Californie) qui utilise un capteur optique particulier dénommé “Synthetic Aperture Optics” qui permet d’améliorer la résolution d’analyse de séquençage par fluorescence. Le processus consiste à éclairer la zone à capter avec des formes lumineuses différentes et à reconstituer avec ces images successives prises avec un capteur donné une image de résolution supérieure. C’est bien un fournisseur de technologie et pas une société à même de fournir un système de séquençage clé en main qui n’est qu’une des applications de son système.
Il faut signaler ici que cette série d’articles n’examine pas toute une catégorie de machines de tests d’ADN très largement diffusée dans les laboratoires. Celles-ci servent non pas à séquencer les génomes mais à identifier des parties d’ADN déjà connues et avec des marqueurs. Par exemple, des gènes ou des gènes modifiés. C’est dans ce domaine que l’on trouve les “DNA Arrays” et “DNA microarrays” (puces à ADN) qui permettent de tester un ADN dans des matrices de tests où chaque test va correspondre à un marqueur donnée, le plus souvent à un gène. On va aussi évaluer l’expression des gènes, à savoir identifier les gènes opérants d’un organisme vivant. On le fait au travers de tests sur les ARN messagers d’un échantillon pour voir quels sont les gènes de l’ADN qui sont transposés en ARNm, qui produisent ensuite les protéines à l’aide des ribosomes. Autre application : détecter les “polymorphismes” génériques, à savoir les variations des gènes par rapport à une référence d’espèce. Cela sert aussi bien dans la santé que dans la police scientifique.
Mais nombre de ces machines exploitent des processus biochimiques et des capteurs voisins de ceux que l’on trouve dans les machines de séquençage. Ces machines sont plus spécialisées. Leur traitement est en général plus rapide que celui du séquençage et aussi moins couteux. Il y a aussi beaucoup moins de calculs numériques dans le processus car il y a moins de données à récupérer et on ne cherche généralement pas à reconstituer le puzzle des suites de bases de l’ADN récoltées en petits morceaux.
Cependant, le marché pourrait évoluer pour certaines applications en direction des machines de séquençage. Aujourd’hui, séquencer tout un génome ne sert à rien si l’on cherche à identifier juste quelques gènes correspondant à une pathologie donnée. Mais comme le cout du séquençage baisse inexorablement, on risque de voir de plus en plus d’applications se reposer sur celui-ci. Qui peut le plus peut le moins… Mais les tests de biogénétique ne concernent pas que l’ADN lui-même on l’a vu, donc le séquençage ne sera pas la réponse à toutes les questions.
C’en est terminé pour cette partie sur les technologies biologiques et matérielles de séquençage. Il faudrait pouvoir benchmarker ces machines … mais comment ? Qui fait cela ? Pas trouvé.. mais cela va bien arriver un jour.
Etape suivante… nous allons revenir au bon vieux numérique et couvrir le traitement des données de séquençage qui sont plus qu’abondantes. Avec notamment du big data au programme, mais pas seulement !
![]()
![]()
![]()
Reçevez par email les alertes de parution de nouveaux articles :
![]()
![]()
![]()


































 Articles
Articles
Les technologies de séquençage du génome humain – 4 http://t.co/6x3ZQbat #innovation: Les technologies de séquen… http://t.co/ysRu5Wv8
“Les technologies de séquençage du génome humain – 4” de @olivez sur http://t.co/3hAQhatk
Beaucoup d’entreprises US dans cet article, peu d’européens à part UK à moins que j’en oublie. Avez vous croisez d’autres acteurs européens lors de vos recherches ? Comment expliquer cette répartition géographique des entreprises du séquençage ?
Toutes les techniques sur base de nanopores paraissent très prometteuses et font figure d’innovation de rupture par rapport aux méthodes de séquençage précédemment évoquées. Savez vous si cette techno est aussi envisagée pour faire d’autre analyse que du séquençage ADN ? Avez vous eu un aperçu de comment sont fabriqués les protéines et comment sont elles fixées sur les plaques de silicium ?
Bonjour Pierre,
Dans un des articles suivants, je reviendrais sur la structure de cet écosystème et pourquoi l’Europe est assez absente et en particulier la France.
La technologie d’Oxford Nanopore Technologies permet de faire des analyses de caractérisation de protéines.
Pour le reste, je suis un peu sec. Je n’ai pas trouvé de documentation sur les procédés de fabrication des protéines ou lipides divers de ces nanopores lorsqu’ils sont biologiques (vs pur silicium). Ils doivent être sûrement brevetés, donc cela doit pouvoir se retrouver quelque part. A moins que cela ne relève du secret industriel.
Bonjour Olivier,
Je suis tombe sur votre blog en cherchant a voir sur ce qu’il se dit en francais sur le nouvelles techniques de sequencage. Je suis bluffe par votre article tres pousse et tres complet sur toutes ces techniques, surtout en sachant que ce n’est pas votre specialite…bravo!
Comme Pierre je suis assez curieux de savoir pourquoi la France est aussi larguee dans ce domaine, avez vous publie votre article sur les raisons de cette absence d’un marche qui va certainment depasser les 20 milliard de dollars en 2025? Je ne l’ai pas trouve dans vos archives…
Avec trois ans de retard, pour la fabrication des proteines on utilise des proteines transmembraines presentent dans la nature comme l’alpha hemolysine du strptocoque
. Ces proteines s’auto assemblent a travers une membrane faite de polymere artificiel mimant les phospholipide mais plus stables.
J’explique cela dans cette partie de cette série d’articles : http://www.oezratty.net/wordpress/2012/technologies-sequencage-genome-humain-6/. J’ai aussi mis à jour l’état de l’art du séquençage dans cet article plus récent : http://www.oezratty.net/wordpress/2015/derive-exponentielles-3/.
On est en effet largués de ce point de vue. Ce, malgré les travaux réalisés en France, notamment à l’Institut Pasteur et au Généthon, pour contribuer au premier séquençage du génome humain.
La France peut encore se rattraper sur l’impression d’ADN. J’ai découvert cette semaine une startup prometteuse du secteur : DNA Script qui ambitionne de créer une solution technique bien moins coûteuse et plus rapide que les solutions existantes, dominées par les américains, comme chez SGI-DNA.