Dans la partie précédente de cette série sur la renaissance des startups scientifiques lancée fin 2016, j’avais commencé par faire un panorama rapide et probablement très incomplet de leur écosystème en France. Je me tourne à partir de cet épisode sur un panorama thématique de quelques startups scientifiques que j’ai pu rencontrer de près ou de loin et qui m’ont marqué pour une raison ou une autre. La plupart sont françaises mais j’ai intégré diverses startups étrangères et notamment américaines pour créer un éventuellement contraste.
Nous allons commencer par les startups du domaine de la santé. J’évacue volontairement la plupart des startups de la e-santé qui n’exploitent généralement pas un bagage scientifique significatif. L’une des plus connues est probablement Doctolib qui bat des records de croissance et de levée de fonds avec son système de prise de rendez-vous avec les praticiens.
Les startups scientifiques du secteur de la santé peuvent être classifiées grossièrement entre medtechs et biotechs. Les premières planchent sur les outils de diagnostic ou opératoires et les secondes sur les thérapies avec quelques chevauchements entre les deux comme pour les outils liés à la chirurgie ou à l’ophtalmologie.
Ces articles n’ont évidemment la prétention d’inventorier les startups scientifiques de manière exhaustive. Libre d’ailleurs à vous d’en mettre en lumière d’autres par le biais des commentaires de l’article. J’ai pu les identifier dans différents endroits comme dans la conférence Hello Tomorrow organisée à Paris en octobre 2016 par Xavier Duportet et son équipe, au Web Summit à Lisbonne en novembre 2016, chez Scientipôle Initiative ou dans mes déplacements divers, notamment à Lyon fin 2016 et Strasbourg fin janvier 2017. Elles m’ont marqué par les utilisations de leurs solutions et/ou leur histoire.
A chaque fois, j’essaye au passage de formuler des défis scientifiques et d’usages qui permettraient de conquérir de nouveaux marchés en étant plus nettement différencié. Comme d’habitude, un peu à l’instar de ce que je peux faire dans le Rapport du CES, je scanne un grand nombre de sociétés. C’est une approche bottom-up qui me permet parfois d’identifier des patterns. Excusez du double anglicisme ! En français, des points communs, des tendances, des facteurs clés de succès ou d’échecs.
Je me pose en particulier cette question lancinante : qu’est-ce qu’une plateforme ? A quoi ressemblent-elles dans l’univers de la santé ? Idéalement, ce sont des produits assez génériques, avec des consommables pouvant être d’origine tierce-partie comme dans le secteur des analyses du sang. Cela peut aussi être des plateformes logicielles d’analyse de données comme celle de Portable Genomics, centrées sur l’ADN ou d’autres données. Les solutions les plus innovantes qui utilisent des techniques d’intelligence artificielles se différencient le plus souvent par le volume de données qu’elles sont capables de consolider et d’analyser. C’est le prix à payer pour rendre efficaces les algorithmes de machine et deep learning.
A l’instar des startups de l’univers des objets connectés, celles qui sont citées ici sont à cheval entre de nombreuses disciplines : la biologie moléculaire, la génomique, l’imagerie, la microfluidique, l’informatique embarquée, les mathématiques, l’intelligence artificielle, les réseaux et le cloud. Nombre de ces disciplines font appel à des travaux de recherche récents, et d’autres à des savoir-faire technologiques récents. L’un de leurs principaux défis, au-delà du financement, est donc d’assembler des équipes pluridisciplinaires et de bien les faire travailler ensemble.
De plus en plus souvent, une startup innovante, surtout dans les medtechs, devra faire appel aux travaux de recherche de plusieurs laboratoires et pas d’un seul comme on le pratiquait traditionnellement jusqu’à présent. Cela doit pousser à raisonner différemment des modèles d’innovations trop linéaire qui ont court jusqu’à présent avec : projet de recherche, chercheur entrepreneur, accompagnement, adjonction d’une compétence business, développement d’un produit basé sur un seul procédé, vente en OEM/marque blanche ou en marque propre, etc.
Génomique
Ces sociétés proposent des solutions qui couvrent tout ou partie de la chaîne de valeur de l’ADN : le séquençage, qui analyse les bases une par une dans tout ou partie de l’ADN, le génotypage qui caractérise des séquences précises avec des techniques plus rapides et moins coûteuses que le séquençage, la modification d’ADN comme avec les fameux ciseaux enzymatiques d’ADN CRISP-Cas9 et, enfin, la biosynthèse qui vise à produire de l’ADN artificiel, ex-nihilo ou, généralement à partir d’une séquence d’ADN existant.
Les grandes medtechs de la génomique sont à dominante américaine, Illumina en premier, suivi des groupes Thermo Fisher et Pacific Bioscience qui ont acquis de nombreuses startups du secteur du séquençage de l’ADN, du génotypage voir de la biosynthèse d’ADN. Il faut aussi compter avec Synthetic Genomics, la société de Craig Venter spécialisée dans l’ADN de synthèse.
Il y a très peu de sociétés françaises dans ce secteur. La France a raté cette vague comme expliqué dans un article de septembre dernier. A la fois parce que le contexte médical n’y était pas propice mais aussi parce que les financements disponibles ne permettent pas d’aller très loin sans en chercher hors de France. Il y en a tout de même quelques-unes qui s’aventurent dans le séquençage. Les plus nombreuses se situent dans le créneau du génotypage ciblé sur certaines pathologies, notamment dans l’oncologie. Elles font face à une rude concurrence de startups américaines du même domaine comme Acuamark Diagnostics, Grail, Clinical Genomics, CellMax ou encore PrezDiagnosic qui détecte les débuts de la maladie d’Alzheimer. Ce sont des startups qui s’attaquent le plus souvent à des marchés de niche.
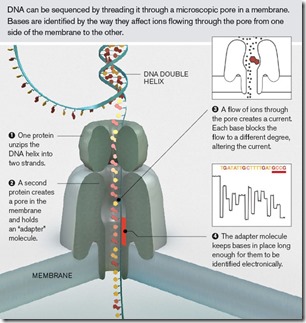
Oxford Nanopore est une startup britannique très intéressante qui a développé la technologie de séquençage de l’ADN et de l’ARN la plus miniaturisée au monde, tenant dans un simple accessoire de smartphone. Avec leur MiniION, on ne séquence pas l’ADN humain en entier avec ses 3 milliards de base mais des gènes ciblés. C’est déjà pas mal ! Cela permet par exemple d’identifier rapidement des pathologies précises. Le procédé est très séduisant sur le papier avec l’exploitation de nombreux nanopores au travers desquels sont entrainés des demi-brins d’ADN ou des brins d’ARN, avec une détection magnétique de chaque base au gré de leur passage dans le pore. La méthode n’est probablement pas sans inconvénients, comme les limitations de la taille des brins d’ADN ou d’ARN analysables. Elle n’en reste pas moins révolutionnaire (voir l’intervention de Clive Brow, son CTO à Hello Tomorrow à Paris en octobre 2016).
Depixus (anciennement Picoseq) est une startup francilienne fondée en 2012 issue du Laboratoire de Physique Statistique de l’ENS Ulm, spécialisée dans l’analyse génétique. Elle propose SIMDEQ, une nouvelle solution pour le séquençage de l’ADN ou d’ARN qui permet d’analyser une molécule unique sans amplification de type PCR et à base de micro-billes magnétiques. Elle peut séquencer ainsi des brins d’ADN de plusieurs milliers de bases. Je les avais vus passer à Scientipole Initiative en 2013. la société a été créée par un Anglais, Gordon Hamilton. Mais cela n’a visiblement pas suffit à lui permettre de lever des fonds significatifs, qui ne sont pas documentés. Pourtant le procédé technique a l’air très intéressant au premier abord.
Stilla Technologies est un finaliste du Grand Prix de l’Innovation de la Ville de Paris 2016. La société a créé Naica System, un système d’analyse génétique à base de PCR (Polymerase Chain Reaction) qui exploite un échantillon biologique après l’avoir disséminé par microfluidique dans jusqu’à 30000 micro-réservoirs. L’ADN de chaque réservoir est répliqué par PCR (un processus chimique et thermique) et analysé ensuite par réaction du résultat à des polynucléotides préinstallés dedans (détails du procédé). Le système permet ainsi de détecter des mutations génétiques responsables de certains cancers ou dans le cadre de diagnostic pré-natal. La détection utilise un capteur photo CCD.
Genomic Vision est une société française créée en 2004 par un ancien chercheur de l’Institut Pasteur, Aaron Bensimon. Elle propose une autre solution de génotypage efficace, reposant sur une technique d’étirage de l’ADN et de dépôt sur un substrat de verre chimiquement traité (vidéo). Elle permet de tests d’analyse de gênes dans le cadre de différentes pathologies d’origine génétique ainsi que dans l’oncologie. La société a démarré récemment la commercialisation aux USA.
Dendris est une startup française créée en 2009 à partir de travaux de recherche brevetés en 2003, basée à Labège près de Toulouse, également positionnée dans le génotypage rapide, à base de DNA arrays [ajouté le 6 mars 2017]. La technique utilise des dendrimères (molécules en forme d’arbres qui se fixent à un support) au phosphore qui permettent de fixer plus facilement les brins d’ADN à analyser et d’obtenir une très bonne précision de la mesure, même à très faible dosage.
Magnomics (Portugal, vue au Web Summit) permet de réaliser des diagnostics in-vitro à base d’extraction d’ADN, d’amplification (PCR) et de détection magnétique. L’appareil permet de détecter des bactéries et des résistances aux antibiotiques à l’aide d’une simple goutte de sang. Les utilisateurs sont aussi bien les médecins que les vétérinaires.
Twist Biosciences est une startup de la Silicon Valley fondée par une française, Emily Leproust, que j’ai eu l’occasion de croiser lors de la conférence Hello Tomorrow où elle intervenait en octobre 2016 à Paris. Emily vient de Lyon après un diplôme d’ingénieur de l’Ecole Supérieure de Chimie Physique Electronique. Elle fait un doctorat sur la chimie de l’ADN à l’Université de Houston puis est embauchée par Agilent où elle travaille pendant 13 ans dans la production d’ADN de synthèse. Elle y créé une première machine adaptée. Elle les quitte en 2013 pour fonder Twist Bioscience à partir d’une idée technologique qu’elle a en tête pour créer une solution encore plus efficace de production d’ADN de synthèse s’appuyant sur des wafers en silicium [Agilent a attaqué Twist en justice début 2016 pour vol de propriété intellectuelle. Twist a récemment contre-attaqué publiquement].
Sa démarche est industrielle : comment produire l’ADN de manière fiable et à grande échelle, avec des brins d’ADN aussi longs que possible ? Sa société a levé $133m et comprend plus de 130 personnes qui se répartissent en trois tiers de compétences: biologie, industrialisation et logiciels. Elle créé ses propres machines de synthèse d’ADN qu’elle utilise dans un modèle de service, qui peuvent produire des brins d’ADN de 1800 bases.
L’ADN de synthèse résout des problèmes dans de nombreux secteurs : pour produire du riz plus riche en vitamine A, de la vanille artificielle, de l’artémisinine synthétique avec de la levure au lieu de plantations d’armoise pour traiter la malaria, pour de l’immunothérapie anticancéreuse ciblée, pour traiter efficacement la biomasse, pour produire de la corne de Rhinocéros synthétique (via Pembient) et du squalène artificiel au lieu de tuer rhinocéros et requins. Enfin, l’ADN créé peut aussi stocker de l’information. Twist travaille ainsi avec Microsoft à qui il a fournit des millions de bases d’ADN en séquence et qui a été décodée sans erreur. Son ambition ? Devenir leader mondial du secteur et concurrencer notamment SGI-DNA, la société de Craig Venter qui est positionnée sur le même marché ! Pas fou, le leader mondial du séquençage de l’ADN, l’Américain Illumina, est l’un des investisseurs de Twist ! (voir l’intervention d’Emily Leproust à Hello Tomorrow)
DNA Script est une startup parisienne créée par Thomas Ybert, Polytechnicien, et Sylvain Gariel, ingénieur du Corps des Mines. Leur but est de produire de l’ADN synthétique en utilisant une méthode de synthèse utilisant une catalyse enzymatique qui permet de réduire de plusieurs ordres de grandeur le cout et la durée de cette production et au passage, d’en améliorer la fiabilité. Le procédé repose comme il se doit sur des brevets déjà déposés. La société collabore avec l’Institut Pasteur et l’ESPCI Paris. C’est un concurrent de l’américain Molecular Assemblies qui utilise un procédé voisin.
Leur approche est long terme : le procédé demande encore de la mise au point pour être industrialisé au-delà de quelques bases d’ADN. Leur produit ne sera opérationnel qu’en 2019 dans le meilleur des cas. Ils sont devenus lauréats de Scientipôle Initiative en 2015 et ont déjà gagné le concours iLAB en 2016. C’est un projet aussi ambitieux que celui de Twist Bioscience, mais lancé à partir de la France. Leurs histoires parallèles seront intéressantes à comparer d’ici quelques années ! Il serait bon en tout cas qu’ils bénéficient d’un bon niveau de financement pour industrialiser leur procédé, quitte pour ce faire, à mettre un pied aux USA ou ailleurs !
Analyses biologiques
Un second domaine des medtechs donne lieu à la création d’un grand nombre de startups, dont certaines sont listées dans le dernier Rapport du CES 2017, pages 258 à 262 : l’analyse biologique du sang et, dans la lignée, celle de l’urine. Les plus nombreuses sont celles qui s’intéressent à la mesure continue et non invasive du glucose dans le sang pour les diabétiques. La raison en est que le diabète est la première maladie chronique et monde et que l’on n’en guérit pas, sauf dans quelques cas de diabète type 2, dit insulinorésistant. En conséquence, le marché de la meure de la glycémie qui nécessite généralement des consommables est très attirant pour les entrepreneurs.
Nous avons ensuite les startups qui ambitionnent de mesurer un maximum de paramètres au-delà du glucose : bilan lipidique, bilan rénal, fonctionnement de la thyroïde avec la TSH, bilan prostatique avec la PSA, bilan des transaminases qui détecte des lésions dans le foie, le cœur, les reins et les muscles, bilans hormonaux divers comme en gynécologie, mesure du taux de plaquettes, bilan lymphatique par comptage des différentes sortes de globules blancs, etc. Sauf pour ces derniers, ces paramètres se situent niveau moléculaire. D’autres tests peuvent avoir lieu qui concernent la génomique, vus auparavant, ou l’expression de gênes, via la détection d’ARN issue de la transcription de l’ADN ou de protéines ciblées. Ils servent surtout à détecter des maladies génétiques avant la procréation, sur les fœtus, ou des cancers émergents.
Les techniques d’analyse biologique du sang sont aussi variées que sa composition. L’électrochimie est notamment utilisée pour la mesure de la glycémie dans le sang. Elle consiste à appliquer un réactif sur le sang puis à mesurer la conductivité électrique du sang ainsi traité, le plus souvent dans de petites languettes alimentées par une goutte de sans avec un procédé de microfluidique. La mesure doit au minimum tenir compte de la température ambiante. Elle est aussi visiblement utilisée par l’Américain Cue (vidéo) et son appareil à capteurs interchangeables.
L’électrophorèse est utilisée pour l’analyse de certaines protéines dans le sang et dans les urines, comme l’albumine. Elle s’appuie sur l’application d’un champ électrique aux ions d’une solution aqueuse ou sur un gel. Ces ions se déplacent en fonction de leur taille. La technique est à ce jour difficile à miniaturiser. Elle a servi pendant longtemps au séquençage de l’ADN, via la méthode de Sanger, qui est maintenant dépassée par les techniques de séquençage de seconde et troisième génération.
On peut aussi analyser la réaction du sang à un traitement par fluorescence et analyse via un capteur photo qui va compter les cellules réagissant au traitement, ce que propose le grenoblois Avalun. La spectroscopie dans le visible ou l’infrarouge est de son côté utilisée pour détecter la présence de certains composants dans la matière inerte ou vivante, comme Scio pour détecter des engrais chimiques ou des allergènes. La spectroscopie vibratoire est utilisée par la startup américaine Cor pour l’analyse sanguine. Il existe en fait une vingtaine de méthodes différentes de spectroscopie !
La quasi-totalité de ces techniques d’examens passe par l’usage d’un consommable. C’est à la fois une contrainte technique et un impératif business permettant de générer un revenu récurrent. Je ne connais pas de technique d’analyse ne nécessitant pas de consommable qui aurait été bloquée pour cette dernière raison. La mesure non invasive du glucose dans le sang est dans les tablettes de pas mal de startups et grandes entreprises (Alphabet + Roche) mais n’aboutit pas encore car elle prend du temps à être mise au point.
Examinons donc quelques-unes de ces sociétés :
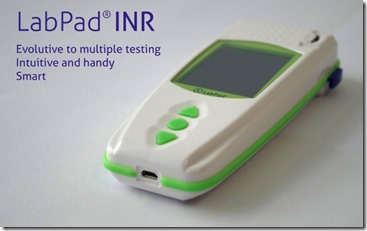
Avalun est une startup issue du CEA-LETI que j’avais rencontrée à à Grenoble en 2015 (compte-rendu). Elle propose un lecteur portable d’analyses biologiques, le LabPad, qui exploite un capteur de goutte de sang utilisant de la microfluidique et deux capteurs d’imagerie CMOS. Le premier sert à faire de la microscopie de l’échantillon pour le comptage de cellules réagissant à un réactif chimique ou à une source de lumière LED, et l’autre pour récupérer le code barre de la languette de consommable. Ce LabPad peut servir à réaliser plusieurs tests biologiques différents, via sa gamme de consommables qui comportent un réactif et un code barre uniques. La première application est la mesure du taux de coagulation du sang pour les malades ayant eu un AVC et sous traitement anticoagulant. Suivront la mesure de la créatinine qui évalue la fonction rénale, l’hémoglobine, les bilans lipidiques, la qualification d’une grippe et la mesure des fibrinogènes, ces fibres du sang qui provoquent la coagulation. Les essais cliniques sont en cours depuis 2014. Le produit est toujours en cours d’homologation. La startup a été créée fin 2013. Elle cherche toujours à lever des fonds pour accélérer son industrialisation. On n’est ici pas très loin du mythique Tricorder capable d’identifier différentes pathologies via diverses analyses sanguines. On a une base de plateforme matérielle, les cconsommables pouvant en théorie provenir de tierces parties.
Brainshake est une startup finlandaise qui fournit une plateforme d’analyse de marqueurs de maladies chroniques avec plus de 220 paramètres mesurés dont le cholestérol, les triglycérides, les lipides et les acides aminés. Leur procédé dénommé “Serum NMR Metabolomics Platform” est fourni sous la forme de service. On envoie un échantillon de sang et la société fournit les résultats via des moyens numériques. C’est une méthode voisine de celle de la très controversée Theranos, qu’elle n’avait pas réussi à mettre au point. La méthode utilisée par Brainshake est un bien documentée avec la liste des appareils de mesure du marché qui sont exploités. C’est du matériel de laboratoire qui n’est pas du tout miniaturisé. C’est donc un simple business de service optimisé.
Aterovax est une entreprise française créée en 2006 qui a développé AteroDx Activity, un test sanguin mesurant l’enzyme phospholipase A2, qui est un bio-marqueur de prédiction des accidents cardiovasculaires. La société a levé un montant non communiqué, probablement de l’ordre de 1,5 m€qui était annoncé en 2013 mais n’aurai été levé qu’en 2015. Le sujet traité est intéressant mais on est évidemment loin du mythique tricorder car la startup s’attaque à la mesure d’un seul paramètre très précis.
Biosynex est une PME alsacienne spécialisée dans la création de tests de diagnostics médicaux à lecture rapide pour les professionnels qui sont utilisés pour la détection de pathologies tropicales, de maladies auto-immunes et pour des maladies infectieuses. Ils servent surtout à accélérer la prise en charge de patients en milieu hospitalier et d’urgences. L’offre comprend notamment le test ImmunoQuinck Dengue détectant le virus de la dengue à partir de quelques gouttes de sang. Le test ImmunoQuick Syphilis détecte en cinq minutes les anticorps responsables de la Syphilis. La société a même fait l’acquisition des groupes allemands Dima Diagnostik et Sensogen Deutschland. La société a réussi une augmentation de capital en levant 3m€ fin 2016, s’ajoutant à plus de 2m€ levés les années précédentes.
BioSIMS est une startup de Rouen qui analyse des protéines régulatrices de l’activité cellulaire et des protéines du plasma avec sa technologie DigiPLEX. Elle détecte dans le sang les biomarqueurs secrétés par les organes malades.
PKVitality est une spin-off du groupe PK Paris qui s’est spécialisée dans la mesure non invasive de la glycémie (pour les diabétiques) et du lactose (pour les sportifs) avec un capteur intégré dans une montre récupérant sans douleur du liquide interstitiel, juste sous le derme de la peau. La solution est décrite dans le Rapport du CES 2017. J’ai même eu le plaisir de faire intervenir Luc Pierart (CEO) et Armelle Plassart (CMO) dans la conférence le retour du CES 2017 organisée par Cap Digital et Systematic le 25 janvier 2017 à Paris. C’est un cas intéressant d’une société qui s’est diversifiée à partir d’un domaine qui n’avait rien à voir, celui du stockage mobile. Ils ne sont cependant pas seuls sur ce marché, avec l’Américain Genteel et l’Anglais Nemaura Medical qui exploitent visiblement le même procédé technique qu’eux.
Sano Intelligence est une autre startup, basée à San Francisco, qui veut permettre la mesure de la glycémie en temps réel pour les diabétiques. La startup a déjà levé $31m ! Mais elle ne communique absolument pas sur la méthode d’analyse, ce qui est un peu louche. On imagine qu’ils utilisent aussi l’analyse du liquide interstitiel comme PK Vitality.
L-See est une startup corse qui exposait au CES 2016 et propose un outil d’analyse du métabolisme des lipides et permet de caractériser l’évolution de la masse graisseuse et musculaire, avec des applications centrées sur le sport. Ils utilisent un procédé d’électrochimie appliquée à un marqueur du métabolisme de la transformation des lipides en glucides. C’est une approche intéressante, mais difficile à généraliser à tous les marqueurs biologiques du sang.
Sencet est un projet de recherche CNRS/LIMMS-SMMILE basé à Lille qui planche sur la détection de cellules cancéreuses dans le sang avec un système dénommé Silicon Nanotweezer issu de travaux de recherche du CNRS et de l’université de Tokyo. Ce système nanoscopique à base de silicium est capable d’immobiliser les cellules analysées une par une et de mesurer simultanément leur résistance mécanique et électrique avec un nano-actuateur. Les données récupérées permettent de caractériser les cellules cancéreuses. Le projet est en cours de transformation en startup.
Fluigent est une PME française spécialisée dans la microfluidique, un domaine technique critique pour les analyses biologiques permettant d’identifier des cellules rares. C’est une technologie de base qui peut être commercialisée sous forme de brevets ou en OEM auprès de nombreux fabricants de dispositifs médicaux d’analyses biologiques.
Tricorders
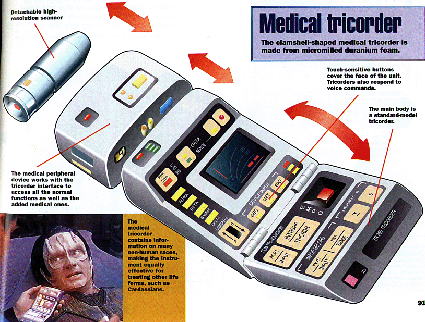
Le but recherché par les entrepreneurs du secteur de l’analyse biologique est d’aboutir un jour à la création d’un appareil unique capable de réaliser d’une seule passe ou en plusieurs passes un nombre d’examens aussi grand que possible, et si possible, à domicile. Il relève de l’appellation de “tricorder”, l’appareil mythique utilisé par le Docteur McCoy dans Star Trek et qui est capable d’analyser l’environnement et le corps humain d’un seul coup.
Le tricorder idéal serait aussi capable de faire de l’imagerie médicale intégrée en 3D, via un scanner amovible (cf l’illustration ci-dessus). C’est un Graal bien plus lointain que celui de l’analyse biologique intégrée compte-tenu de l’état de l’art de l’imagerie médicale non invasive.
Qualcomm a lancé en 2012 un prix Tricorder XPrize doté de $10m pour financer quatre projets de ce type. Il y avait au départ 300 équipes candidates. Le concours quinquennal a éliminé les candidats étape par étape. Il reste une demi douzaine de candidats en lice dont deux finalistes qui seront départagé d’ici mi 2017 pour gagner une dotation de $6m pour le premier, $2M pour le second, et $1m pour celle des autres équipes capable de mesurer le plus grand nombre de paramètres vitaux.
L’objectif est de permettre la détection d’une grosse douzaine de syndromes avec un appareil aussi intégré et miniaturisé que possible, exploitable par des médecins voir par les patients eux-mêmes : l’anémie, diverses pathologies cardio-respiratoires dont l’hypertension, les anomalies lipidiques, le diabète, les pathologies ORL, l’apnée du sommeil, les infections urinaires, le SIDA, les troubles de la thyroïde et les mélanomes. Il faut au minimum que l’appareil puisse mesurer la tension artérielle, la saturation en oxygène du sang, le rythme respiratoire et la température, tout ceci faisant partie de l’état de l’art actuel. A ce jour, les finalistes sont encore loin du compte. A ce que l’on peut en voir, ils se contentent d’assembler pour l’instant de manière plus ou moins élégante diverses techniques existantes.
L’enjeu principal est, comme ce qu’a fait Scio dans la spectroscopie infrarouge, de miniaturiser à l’extrême et de manière modulaire des techniques d’analyses de laboratoires. Il est par exemple difficile de miniaturiser des dispositifs d’électrophorèse. L’autre porte sur la durée des tests. Certaines sociétés contournent ce problème en faisant l’analyse de paramètres divers, issus notamment de mesures non invasives, et en faisant des calcules de corrélations via le big data et le machine learning avec les paramètres biologiques. Parfois, cela relève de l’escroquerie ou du charlatanisme comme la technique d’électro-acupuncture du croatien Vitastiq évoqué dans le Rapport du CES 2017, page 263.
Voici donc les équipes en lice et leur solution de tricorders à commencer par les deux équipes finalistes pour le prix de $6m :
L’équipe Taïwanaise Dynamical Biomarkers Group a créé un système à base de trois modules : un moniteur de signes vitaux, un système de test de l’urine et un imageur baptisé Smart Scope. Ils sont tous reliés à un smartphone. Ils sont destinés à être utilisés directement par les patients, qui seront guidés par l’application mobile associée qui exploite, comme il se doit, des techniques d’intelligence artificielles non précisées. Les procédés utilisés pour les analyses biologiques ne sont malheureusement pas documentés. Le packaging a l’air encore un peu trop volumineux pour une application grand public. Le projet a été accompagné financièrement par HTC, plus connu pour ses smartphones et son casque de VR Vive.
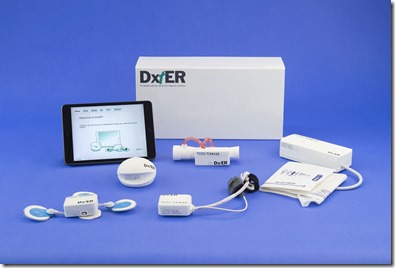
L’équipe américaine Basil Leaf Technologies (équipe Final Frontier Medical Devices) a créé la solution DxtER qui s’appuie essentiellement sur une application tablette dotée d’une intelligence artificielle capable d’exploiter les données issues de plusieurs capteurs modulaires, la plupart provenant du marché, dont un capteur d’ECG, un oxymètre de doigt qui a l’air de faire un dosage sans prélèvement de sang de l’hémoglobine, des globules blancs et même de la glycémie et un stéthoscope. Le projet est basé en Pennsylvanie.
Les autres équipes sont aussi intéressantes et leur solution parfois bien plus disruptive que les sages solutions des deux finalistes :
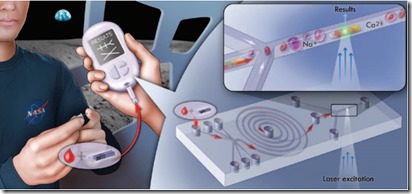
L’américain DNA Medicine Institute (DMI) a créé le rHEALTH (Handheld Electrolyte and Lab Technology for Humans), un appareil de mesure biologique du sang à base d’un système avancé de microfluidique et de fluorimétrie laser qui m’a l’air plus intéressant que les capteurs des deux finalistes. Le système est capable de décompter les cellules réagissant à des réactifs chimiques. Cela lui permet de faire des dosages de certaines protéines et autres marqueurs biologiques. Il n’a pas l’air d’être bien éloigné du capteur modulaire du français Avalun. Cet appareil est le résultat de recherches menées avec la NASA et dans le cadre d’un financement SBIR, équivalent des financements de projets de recherche par l’ANR en France. Il visait au départ à créer un tel appareil pour les analyses biologiques dans la station spatiale internationale. L’appareil fonctionne déjà en gravité zéro ! A ce jour, il est capable de mener 22 tests différents. La partie microfluidique de l’appareil n’est pas un consommable. Seuls les agents chimiques le sont. L’équipe est basée à Cambridge, à côté de Boston.
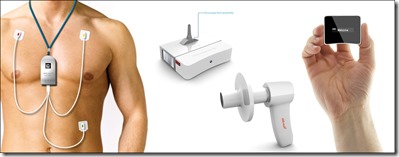
L’équipe UK-USA Intelesens-Scanadu propose une solution qui a l’air de rassembler un capteur d’ECG d’Intelesens, basé à Belfast en Irlande du Nord, le scanner de l’américain Scanadu et les outils de laboratoire de l’anglais Randox. Scanadu est une startup lancée en 2011 et installée au Ames Research Center de la NASA à côté de Palo Alto, là-même où est se situent les locaux de la Singularity University. Son scanner Scanadu Scout se positionne sur la tempe et mesure la pression sanguine, la température, le pouls, la tension et la saturation en oxygène du sang, visiblement grâce à un capteur infrarouge. Le ScanaFlu est un autre capteur, biologique, capable de détecteur des symptômes gripaux en exploitant la salive. Il détecte les streptocoques, la grippe A et B, les adénovirus et le virus respiratoire syncytial qui affecte les jeunes enfants. Enfin, Scanadu a aussi un test d’urine à base de languette réactive dont les résultats colorés sont analysés par la caméra d’un smartphone en lieu et place d’un contrôle visuel habituel. Après un démarrage sur IndieGogo en 2013, la société a levé en tout $56m. Le Scout a terminé son essai clinique fin 2016 et la FDA n’a pas approuvé sa mise sur le marché, demandant à analyser les résultats de l’étude. Et les fondateurs de Scanadu ont quitté l’entreprise. Donc, c’est mal parti !
L’équipe indienne Danvantri propose un dispositif comprenant un capteur d’ECG, un capteur de tension, température, oxymètre de pouls, complété d’un analyseur de sang non précisé. Tant que l’on ne peut pas savoir comment il fonctionne, impossible d’indiquer s’il y a une réelle nouveauté technologique dans l’ensemble.
L’équipe américaine ClouDX exploitait des capteurs de l’allemand Biosign dont leur moniteur cardiaque Pulsewave. Leur solution était centrée sur la détection de pathologies cardio-vasculaires. Manque de bol, Biosign a fermé boutique mi 2016 !
Pour faire un véritable tour de l’état de l’art, il faudrait en fait analyser les solutions proposées par le 300 candidats. Certains peuvent être éliminés du concours car ils ne sont pas prêts mais tout en ayant une solution techniquement intéressante.
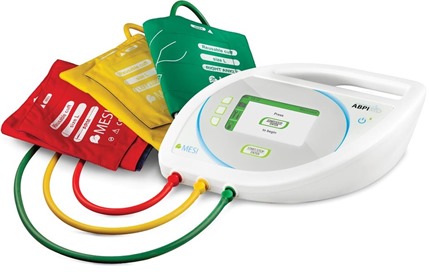
L’un de ces candidats était l’équipe slovène MESI Simplifying diagnostics qui a créé un capteur ne répondant pas aux critères de sélection du Qualcomm XPrize mais qui est néanmoins intéressant, l’APBI MD. C’est un système de mesure de la tension artérielle qui se place sur les deux mains et les deux pieds et qui détecte les problèmes de circulation artérielle périphérique.
Qualcomm n’est pas le seul à encourager la chasse au tricorder. Nokia est aussi de la partie avec son Sensing XChallenge qui s’est conclu en 2014. Le gagnant de ce concours, plus chichement doté, de $525K pour le gagnant, était DNA Medicine Institute, que nous avons déjà cité car il était l’un des finalistes du concours de Qualcomm annoncé en décembre 2016. Les autres finalistes comprenaient notamment :
Golden Gopher Magnetic Biosensing de l’Université du Minnesota et son capteur Z-Lab utilisant la magnétorésistance, exploitant la technologie de la startup Zepto qui elle-même s’appuie sur les travaux du français Albert Fert, lauréat du Prix Nobel de Physique en 2007 pour ses travaux dans la magnétorésistance géante, aussi exploitée dans les disques durs. Les analyses biologiques utilisant la magnétorésistance consistent à utiliser des marqueurs magnéto-résistants qui se combinent avec les cibles à mesurer. Un capteur détecte alors la présence de ces marqueurs. Ce genre de technique est utile pour mesurer des bio-marqueurs qui sont à très faible dosage, là où les techniques optiques à base de fluorescence sont trop imprécises (source).
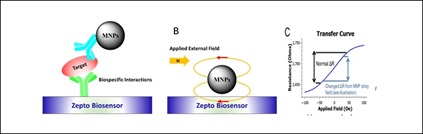
Eigen Lifescience (vidéo) et leur capteur magnétique qui détecte les maladies infectieuses, visiblement, avec un procédé voisin de celui des précédents. Il existe d’ailleurs une société française également positionnée sur l’exploitation de la détection de particules magnétiques par fluorescence pour les analyses biologiques, Immunomag MagIA, basée à Grenoble et issue de l’Institut Néel spécialisé dans la physique des matériaux. Cette dernière société est surtout positionnée sur les tests réalisés par des patients dans le cadre de tests cliniques.
Le français Archimej Technology et son Beta-Bioled, un analyseur de sang portable destiné aux patients mesurant plusieurs paramètres avec une simple goutte de sang, y compris le bilan lipidique, la fonction rénale et hépatique et d’autres marqueurs (vidéo). Le procédé utilise de la spectroscopie multi-spectrale exploitant une matrice de LEDs et une optique de collimation propriétaire qui combine les faisceaux lumineux issus de chaque LED pour illuminer un seul capteur. L’appareil utilise des languettes réactives exploitant le principe de la micro-fluidique, similaire aux lecteurs de glycémie. Une seule goutte de sang permet d’analyser plusieurs biomarqueurs. La languette intègre et automatise tout le processus préalable à l’analyse : centrifugation, mélange avec réactifs chimiques et transfert jusqu’aux zones de lecture. La société prévoit d’aborder en premier lieu la prévention et le suivi d’insuffisances rénales. Les phases de validation cliniques et règlementaires démarrent cette année (2017) pour un début de commercialisation entre 2018 et 2019.
Le spectromètre d’Atoptix qui est un accessoire de smartphone (vidéo). C’est une sorte de variante du capteur de Scio, qui est aussi centré sur le proche-infrarouge, mais sur un autre domaine applicatif. Il est placé contre une artère du bras et mesure les paramètres sanguins de manière non invasive. Cela a l’air intéressant, mais les usages limités n’ont font pas pour autant un tricorder généraliste !
C’en est terminé pour ce petit tour d’horizon des startups de la génomique et des analyses biologiques. On peut y constater la grande diversité des méthodes d’analyses biologiques, de l’importance de la partie logicielle et données de ces outils, surtout lorsqu’ils sont combinés entre eux et des difficultés qui subsistent à créer des appareils et plateformes aussi génériques que possibles. C’est une voie de progrès et d’innovations qui reste à explorer et qui créé autant d’opportunités de rupture pour les entrepreneurs et, collectivement, pour l’écosystème d’innovations français !
La méthode utilisée par Qualcomm pour dynamiser le marché à la quête du tricorder est à retenir pour faire évoluer nos pratiques de financement de l’innovation. Leur prix est doté au total de $10m qui ne sont pas grand chose au regard des investissements dans l’innovation en France. N’importe quel appel à projet des PIA 1, 2 et 3 (Programmes d’Investissements d’Avenir) est doté de plusieurs fois cette somme. Avons-nous un plan de tricorder en France ? Je n’en ai pas l’impression. Pourtant, les compétences existent et de manière disparates pour plancher sur un tel projet.
Comme je l’évoquais dans une autre série de posts récents, au sujet des stratégies industrielles de l’Etat, un plan d’innovation n’a de sens que s’il ambitionne de faire significativement avancer l’état de l’art des secteurs financés. Cela ne sert à rien de faire un plan “objets connectés” ou “big data” ou “intelligence artificielle” si l’objectif n’est pas de faire radicalement mieux que l’existant, voir même de se lancer dans une mission qui parait impossible à réaliser au présent. On appelle cela un “moonshot”. Formuler les plans stratégiques sous forme de défis d’usages et technologiques est une belle démarche que les pouvoir publics pourraient adopter, en s’associant évidemment avec une grande diversité de compétences issues de la société civile.
_________________________
Dans l’article suivant de cette série, nous nous pencherons sur les startups de l’imagerie médicale et des medtechs opératoires. Dans celui qui suivra, nous passerons aux logiciels medtech (hors e-santé) et aux biotechs.
![]()
![]()
![]()
Reçevez par email les alertes de parution de nouveaux articles :
![]()
![]()
![]()



































 Articles
Articles
Olivier tu ne nous laisses pas reprendre notre souffle après la publication récente de ton rapport sur le dernier CES 🙂
Je me pose juste une question concernant ce “capteur intégré dans une montre récupérant sans douleur du liquide interstitiel, juste sous le derme de la peau.”
Comment une piqure peut elle ne pas faire mal ? Quid de la fiabilité d’une telle mesure face à la complexité d’analyse des résultats généralement ?
Ce genre d’outils ne pourra rencontrer un succès commercial que si ils sont individuellement validés par les médecins. Non ?
La piqûre ne fait pas de mal car les aiguilles ne rentrent pas assez profond sous la peau pour taper sur les nerfs. On les sent à peine, je l’ai testé.
Pour ce qui est de la fiabilité, je ne sais pas trop. C’est une mesure indirecte. Il doit y avoir une courbe de corrélation entre la glycémie mesurée sous la peau et celle du sang. Reste à savoir si cette corrélation est la même pour tous les patients et ne nécessite pas de calibrage. Ils ont du tester la chose sur plusieurs patients avant de se lancer. Et comme je l’indique dans le rapport, il y a trois autres sociétés dans le monde qui utilisent ce procédé d’une manière ou d’une autre.
Pour le vendre, il faut obtenir l’agrément dispositif médical des autorités de santé des pays/régions ciblées. Ensuite, il faut qu’il soit recommandé par les diabétologues, comme pour tout produit de ce genre. Enfin, il faut l’agrément des tiers payants (CPAM, mutuelles, …) pour qu’il soit remboursé. C’est le lot commun de toutes les medtechs. Long et fastidieux.
Merci pour nous avoir mentionné Olivier.
Petite rectification et commentaires.
Portable Genomics est maintenant une plateforme mobile, personnelle, de collecte de données de santé, dont les données génomiques associées aux données médicales, comportement, lifestyle et iOT.
C’est une première phase pour nous, s’assurer que ces données personnelles de santé soient captées par les utilisateurs/patients, plutôt que par des silos institutionnels ou commerciaux. Quoi de mieux que de controller de façon totalement privée ses propres données, à partir de son mobile et de son nuage personnel.
Ensuite, pour fournir de l’intelligence sur la base de données génomiques, il faut accéder aux données, il faut que les utilisateurs partagent l’accès à leurs données, et pas seulement celles de leur génome, il faut tout le reste, le médical, lifestyle …etc, il faut que la science y ait accès, il faut que la pharma y ait accès. C’est ca la Precision Medicine.
Nous avons bâtit une infrastructure d’accès et partage de données qui va permettre aux utilisateurs de partager lorsqu’ils le souhaitent, avec qui ils veulent, et lorsque monétisation des données il y aura, nous partagerons les revenus avec les utilisateurs.
Portable Genomics est donc devenue une plateforme de collecte, de partage et de monétisation de données personnelles de santé, y compris données génomiques, avec un nouveau type de sharing revenue model. Nous sommes en beta avec plusieurs groupes de patients, en oncologie ou ayant des maladies génétiques, et avec eux, nous parlons à l’industrie pharmaceutique pour accélérer recherche et découverte de nouveaux médicaments.
Ce qui m’amènent à mes commentaires:
Car la génomique aujourd’hui, en tout cas ce qu’il se passe ici aux USA, est passée dans un nouveau stade industriel, celui de la monétisation des données auprès de l’industrie pharmaceutique. Rock Health avait déjà rapporté l’été dernier que l’industrie de la génomique (à l’exception du fournisseur quasi monopolistique qu’est Illumina), toutes ces boites qui fournissent des tests, services analytiques ou autres, celles qui font vraiment du revenu sont celles qui vendent les data générées à la pharma !!!
Avec maintenant Illumina qui peut fournir des réactifs pour un génome à $100 pourquoi s’embêter à trouver une techno qui le fera passer à $10 voir $1 ? Les efforts de R&D nécessaires sont maintenant trop couteux, et ensuite quel business model appliquer derrière une techno qu’on va vendre moins de $10? Ca se complique.
L’industrie de la génomique préfère gagner peu sur la techno et les services, mais par contre revendre les données $10,000 à $20,000 (cf deal entre 23andMe et Genentech, pour ne citer que celui-la), et au passage, se foutre complètement des consommateurs/patients, qui au passage ne touchent rien, ils ont meme payé pour btenir des data qui se sont revendues dans leur dos. La beauté cruelle des business model d’aujourd’hui.
Mais il y a un hic à l’horizon: le consommateur a senti l’arnaque, et les réglementations sur “privacy & ownership” des data se durcissent, voir HIPAA et CLIA Rules, et sont en faveur des consommateurs. Oops, L’industrie va avoir plus de mal à convaincre le grand public de partager ses données sans partager les bénéfices financiers. Surtout que même à l’heure du génome à $100, les patients atteints de cancer payent encore aujourd’hui un test de susceptibilité génétique de 20 à 70 gènes, $3000, et un profil génomique tumoral $6000. Et L’industrie de la génomique voudrait que ces patients partagent leurs données gratuitement. No way!
Donc ce qu’il se passe en génomique se focalise maintenant surtout sur les business modèles plus que la techno. Bien sur, les services analytiques autour de la génomique ont de beaux jours devant eux, mais il faudra résoudre le business modèle first, car de la en découlera le comment les data vont circuler, qui y aura accès, et comment le patient, qui finalement est le plus concerné par cette affaire, gardera le contrôle lors d’un processus qui doit être complètement transparent. Et on est encore loin du compte.
Car aujourd’hui tu ne partages pas ton génome comme une page Facebook. C’est bcp d’information sur soi, sur ses ancêtres, sur ses enfants. Et qui sait ce que les biohackers de dans 10 ans pourront faire de génomes publiés en open access aujourd’hui. Avec CRISPR et DNA synthesis, ils vont se retrouver avec leurs clones dans la rue. Sans rien avoir vu venir.
Merci pour l’insight !
Je note que la donnée qui a de la valeur est le croisement entre le génome et le phénotype, d’où l’intérêt de collecter toutes les données de santé et pas seulement celles du génome.
Merci Olivier pour cette excellente synthèse des startups de la génomique et analyse bio. Pour être impliqué dans une medtech, je confirme que le parcours est chaotique, long et parfois douloureux, mais une fois les étapes franchies et les produits validés, le marché vaut largement le coup, en particuliers pour les medtech opératoires (mon cas) et biotech. Hâte de lire ton article sur les medtech et biotech.
A+ Yves
FYI: une petite coquille s’est glissée dans cet article. A priori les 2 finalistes auraient le même nom de produit, ce qui est peu probable 🙂
“L’équipe Taïwanaise Dynamical Biomarkers Group a créé le système DxtER.”
“L’équipe américaine Basil Leaf Technologies (équipe Final Frontier Medical Devices) a créé la solution DxtER “
Merci du feedback. En effet, DxtER est le nom du produit de Basil Leaf et pas celui de Dynamic Biomarkers.
“Startups scientifiques : génomique & analyses biologiques” : panorama & analyse d’@olivez – superbe aussi.
https://t.co/fzvi8XQTV3