Voici la dernière partie de cette série sur le séquençage du génome humain qui est dédiée à son exploitation dans la création de thérapies géniques.
Je vais me focaliser ici sur ce que fait le Généthon et les maladies rares d’origine génétiques. La raison ? J’ai eu l’occasion de le visiter en août 2012 ! Mais pas seulement puisqu’on a vu dans la partie précédente que le Généthon avait été central dans l’histoire du séquençage du génome humain en France.
Le Généthon est une sorte de laboratoire de recherche privé créé en 1990 et dont l’essentiel du financement provient de l’AFM-Téléthon qui organise la campagne annuelle de collecte qui a lieu pendant le Téléthon début décembre chaque année depuis 1987. Une collecte qui récupère près de 100m€ par an auprès d’environ 2 millions de donateurs. Soit, l’une des plus grandes opérations de crowdfunding en France, devant les Restos du Cœur (75m€ de ressources annuelles) et Emmaüs (103m€ de ressources, d’origines multiples). Le Généthon gère un budget d’environ 28m€ par an dont l’essentiel – 87% – provient de l’AFM. C’est dans les faits la plus grosse “biotech” de France avec 220 personnes. C’est même un leader mondial des thérapies géniques. Il gère entre autre la plus grande bibliothèque d’ADN et de cellules d’Europe concernant les maladies génétiques humaines avec 236000 échantillons provenant de 73000 individus et couvrant 413 maladies, des échantillons dont l’ADN est séquencé selon les besoins.
L’AFM-Téléthon a ceci de particulier que c’est l’association de personnes affectées de pathologies la mieux organisée et la mieux financée en France. Et surtout, elle a développé son propre agenda scientifique avec une obsession clé, au-delà d’un plan d’action pour améliorer les conditions de vie des malades : guérir les myopathies et par extension, s’attaquer aux plus de 6000 maladies rares qui affectent, en France, 3 millions de personnes. Il se trouve que les maladies rares sont généralement d’origine génétique et faiblement influencées par l’environnement ou le mode de vie, contrairement à un grand nombre de cancers ou de pathologies cardiaques. L’action scientifique passe à la fois par le financement de projets de recherche, le plus souvent de laboratoires publics et en complément des (maigres) financements publics et par son propre laboratoire de recherche, le Généthon.
C’était au lancement de l’AFM en 1987 une vision très long terme. Vingt-cinq ans plus tard, l’association n’a pas à rougir des progrès accomplis. Ils ont démarré par une participation active au séquençage du génome humain et surtout à la cartographie des gènes vers 1992-1993. Ils se sont poursuivis avec une recherche effrénée de thérapies géniques. Les premiers résultats sont arrivés sur une trentaine de maladies rares, avec autant d’essais cliniques de lancés, le plus souvent avec succès, dont un qui a concerné les bébé-bulles victimes d’insuffisances en protections immunitaires. En fait, les myopathies ne sont pas les seules pathologies couvertes par les recherches financées par l’AFM.
La bien compliquée dystrophine
Les myopathies se sont retrouvées un peu plus loin dans la roadmap des thérapies géniques financées par l’AFM. Pourquoi donc ?
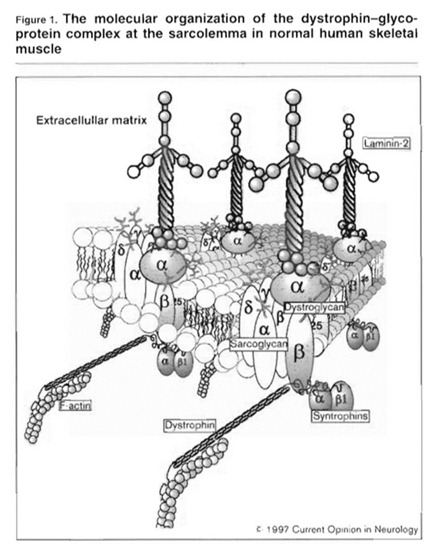
Il faut revenir aux sources. Les myopathies sont généralement liées à une insuffisance de production d’une protéine par un grand nombre de types de cellules musculaires, et notamment celles du cœur, la dystrophine. Elles affectent 1 personne mâle sur 3500. Rappelez-vous l’épisode 1 de cette série : les protéines sont des molécules complexes qui sont des suites d’acides aminés produites à partir de la partie codante des gènes. Il y a environ 30000 gènes dans l’ADN humain, qui servent donc à coder au moins autant de protéines. La dystrophine est une molécule qui en gros sert de liant entre l’intérieur de la cellule musculaire, juste en dessous de la membrane cellulaire, et la laminine, une molécule externe à la cellule qui assure la cohésion intercellulaire du muscle.
Manque de bol, la dystrophine est la protéine la plus complexe des cellules humaines. Son gène, le DMD (pour Dystrophie Musculaire de Duchenne), vient du chromosome X, celui qui est commun aux deux sexes. Il comporte 2,4 millions de bases d’ADN, sur les 3 milliards du total d’un génome humain. Son adresse précise : les paires de bases 31137344 à 33357725 dans ce gène X.
Sur ces 2,4 millions de bases, il y en a seulement 14000 qui sont codantes, à savoir, qui, une fois mises bout à bout par transposition dans l’ARN messager et épissage (élimination des bouts non codants), servent à “encoder” la molécule de la dystrophine. Re-manque de bol, ces 14000 bases sont réparties dans 79 exons (séquences codantes dans le gène) qui sont éparpillés dans les 2,4 millions de bases du gène. Qui plus est, la transposition de ce gène en ARMm est déclenchée par sept promoteurs alternatifs (les promoteurs étant de petites séquences d’ADN qui servent de point de départ à la transposition d’un gène en ARN messager). Ce qui conduit à la production de sept formes différentes de dystrophine selon les cellules. Tout ça, on le sait depuis 1987, l’année de création du Téléthon.
La molécule de la dystrophine fait 427 Kd. Ces kilo-daltons mesurent la masse atomique, un dalton correspondant à un douzième de la masse d’un atome de carbone normal (carbone 12). Vu dans le Genome Browser de l’UCSC, le gène DMD cela donne ceci, ce qui nous avance bien. Chaque petit trait vertical dans les variantes du DMD en haut correspond à un des exons (séquences codantes) du gène. Il faut zoomer dessus pour voir les séquences d’ADN correspondantes et surtout, les polymorphismes associés, les variantes d’un individu à l’autre qui sont identifiées par séquençage.
Les myopathies sont provoquées par un ou plusieurs défauts dans l’expression du gène de la dystrophine avec des bouts d’ADN du gène manquants ou répliqués. Ces modifications d’ADN entrainent soit l’absence complète de production de dystrophine (myopathie de Duchenne) soit une altération de sa production (myopathie de Decker, moins invalidante). Plus de 5000 mutations du gène DMD ont pu être observées grâce au séquençage du génome de personnes atteintes de myopathies. L’essentiel des découvertes sur ces modifications sont intervenues pendant les années 2000 (source), ce qui a permis de mettre en place un diagnostic précis de ces maladies.
Le rôle du séquençage de l’ADN (et de l’ARN) ? C’est un instrument qui fut utile dans toutes ces recherches mais pas le seul. En effet, le séquençage ne permet que de décoder l’ADN. Mais pas de comprendre tout le fonctionnement qui en résulte avec l’expression des gènes, des protéines, l’épigénétique, etc. Le champ de la recherche génétique ne peut pas se contenter du séquençage. Ce n’est qu’un outil parmi d’autres.
Les thérapies géniques
Comment traiter ces pathologies ? Pour mémoire, la myopathie de Duchenne entraine une atrophie des muscles qui invalide les garçons atteints dès leur plus jeune âge. La maladie devient létale car elle atteint le muscle du cœur. L’espérance de vie est de 20 ans.
Des traitements divers inventés dans les années 90 et 2000 ont cependant permis de la rallonger mais pas de manière satisfaisante. Ils ne s’attaquent pas au gène de la dystrophine mais à sa périphérie. Des corticostéroïdes (Prednisone ou Delfazacort) peuvent ainsi ralentir la fragilisation et la destruction des cellules musculaires provoquées par l’absence de dystrophine ou sa présence sous une forme altérée.
Des aminoglycosides (sortes de sucre) ont été testés pendant les années 2000 pour empêcher l’action d’un codon STOP identifié dans les gènes déficients de la dystrophine, concernant 15% des personnes atteintes de DMD. Ce codon est une séquence particulière de trois bases dans l’ADN qui indique à l’ADN polymérase d’arrêter la transposition de l’ADN du gène en ARN messager. Ce codon empêche la protéine d’être construite entièrement après passage via le ribosome qui transcode l’ARNm en protéine. Mais ça ne marche pas. Tout comme tout un tas d’autres traitements testés pendant les années 2000.
Restent les thérapies géniques qui interviennent au niveau du gène lui-même.
Sont testées des “antisense oligomers” qui permettent de modifier le gène défectueux de la dystrophine en supprimant un ou plusieurs exons (zone codante de protéine) qui empêche la protéine de se construire en entier. Cela permet au gène modifié d’engendrer une forme légèrement rétrécie et fonctionnelle de la dystrophine. Des tests de ce genre sont en cours aux USA dont les résultats devraient arriver en 2014.
L’autre méthode consiste à prendre un gène de la dystrophine sain et complet et à en injecter les séquences codantes d’une manière ou d’une autre dans les cellules qui ne l’ont pas et le rendre actif pour qu’il initie la fabrication de dystrophine. C’est évidemment plus facile à énoncer qu’à faire ! Aussi curieux que cela puisse paraitre, la difficulté ne se situe pas tant que cela au niveau des techniques de sélection et découpage du gène dans un ADN sain. Ca, on sait faire. La difficulté consiste plutôt à trouver un véhicule pour transporter le gène sain dans les cellules à traiter et à faire en sorte qu’il y soit accepté et qu’il n’y ait pas de rejet. C’est d’autant plus difficile que ce gène est très long et que la quantité de muscles à traiter est importante et qu’ils sont situés partout dans le corps. Dont un muscle bien particulier qu’il faut traiter sans le brutaliser : le cœur.
On s’appuie pour ce faire sur des “vecteurs”. Ils sont en général de deux sortes : des virus (rendus non pathogènes, exemple ci-dessus) ou des liposomes (sphère en lipides qui entourent le gène et seront digérés par les cellules). Les virus ont soit un ARN (rétrovirus) soit un ADN (adénovirus) capable de greffer leur séquence codante dans les cellules “infectées”. Là encore, nous avons plus d’une douzaine d’années de recherche derrière nous.
Ces méthodes ont été testées pour différentes maladies rares. Ont été expérimentées des thérapies géniques pour traiter l’immunodéficience de “bébés bulle” en 1999, avec 8 enfants guéris sur 10 à l’époque mais avec des effets secondaires. Il y a aussi l’adrénoleukodystrophie, une maladie qui s’apparente à celle de Creutzfeld-Jacobs et détruit la myéline dans le cerveau. Deux enfants ont été traités en France avec succès en 2009 par transplantation du gène responsable, lui aussi présent dans le chromosome X. Idem avec la beta-thalassémie, une maladie génétique qui affecte la production de globules rouges et impose une transfusion sanguine toutes les trois semaines, que l’on sait traiter par thérapie génique depuis 2010 (source 1 et source 2). Autre exemple, enfin, avec des maladies rares de l’œil rendant aveugles. Des chiens aveugles de naissance ont été traités en 2001 et plus récemment en 2011 et 2012 par thérapie génique (achromatopsia, et retinitis Pigmentosa, cf cette vidéo sur YouTube). Mais jusqu’à présent, les thérapies géniques n’ont jamais fait des merveilles à grande échelle.
Sur la DMD (maladie de Duchenne), la méthode sur laquelle planche le Généthon semble être basée sur l’usage d’un système compliqué décrit dans “The Journal of Virology” : “Transfer of the Full-Length Dystrophin-Coding Sequence into Muscle Cells by a Dual High-Capacity Hybrid Viral Vector with Site-Specific Integration Ability“. Avec des vecteurs adénovirus (AAV) hybrides dits intégratifs, où est intégré un double gène DMD humain codant la dystrophine (avec 14 Kbases et non pas les 2,4 mbases d’origine intégrant les zones codantes et non codantes) qui va s’insérer non pas dans le chromosome X mais dans le chromosome 19 dans un endroit précis qui s’appelle le locus AAVS1. Le tout passant par tout un tas d’étapes compliquées que je vous épargne. Sachant que l’administration de ce virus n’est pas orale. Il s’agit d’injections réalisées directement dans les muscles concernés et en milieu hospitalier. C’est plus compliqué qu’un simple comprimé d’aspirine !
Industrialiser la production
Une fois une thérapie génique identifiée, il faut pouvoir la tester et industrialiser sa mise en œuvre. C’est là qu’intervient le troisième étage de la fusée du Généthon, après les recherches sur le génome et sur les thérapies : la production ! Le problème à résoudre est que les thérapies géniques reposent sur de la production ex-vivo de virus que l’on multiplie dans des étuves. Cela dure des heures et n’en produit qu’une très faible quantité au regard du besoin pour traiter les malades. Même à l’étape des tests cliniques.
C’est pourquoi le Généthon s’est lancé dans un projet très ambitieux avec la création du laboratoire Généthon BioProd, construit à Evry, à proximité de son laboratoire de recherche. Le laboratoire a été construit en 2010-2011. Les infrastructures sont actuellement en place et le laboratoire est en cours d’équipement pour un lancement opérationnel d’ici la fin 2012.
Ce labo représentera la plus importante capacité de production au monde de médicaments de thérapie génique. L’imposant bâtiment comprend 5000 m2 dédiés à la bioproduction et au contrôle des produits dont 2500 m2 de laboratoires classés L3 et confinés et quatre suites de production. La capacité de production sera de 20 lots cliniques par an produits avec 800 litres de culture en bioréacteurs pour les vecteurs de type AAV et 100 litres de culture pour les vecteurs de type lentivirus. Ces bioréacteurs, d’origine allemande, sont les plus puissants et rapides qui soient. Les bioréacteurs sont capables de traiter des cellules en suspension et non en dépôt, ce qui permet d’aller vite.
Voici le laboratoire en question :
Au dernier étage, le niveau technique comprend une quinzaine de centrales de traitement de l’air qui renouvèlent tout l’air des salles de production des dizaines de fois par heure et de manière séparée. C’est une sacrée infrastructure.
Et de longs couloirs le long des salles de production où l’on ne peut entrer qu’au travers de sas et proprement équipé. C’est encore un peu vide car toutes les machines ne sont pas encore installées. La production nécessite la mise en place de processus qualité très rigoureux avec séparation des flux en entrée et en sortie dans le laboratoire, confinement des différentes étapes de production, salles blanches, etc. C’est une histoire de qualité et de processus.
Ce laboratoire a représenté 28m€ d’investissements financés à hauteur de 5m€ par l’AFM, 8 m€ par le Conseil Régional de l’Ile de France, 7 m€ par le Conseil Général de l’Essonne et par le parc de recherche Génopole pour 8 m€, qui est lui-même financé par les collectivités locales, l’état et l’AFM.
Au bout du bout : la production des premiers tests cliniques de thérapies géniques pour traiter la myopathie de Duchenne. Les résultats sont attendus pour 2013, 26 ans après la création du Téléthon. Quelle patience, quelle ténacité ! Mais qui auront été payantes au bout du compte !
Mais ce n’est pas tout. Une fois que les tests cliniques auront eu lieu et on l’espère avec succès, il faudra passer aux étapes suivantes. Les équipes du Généthon et de l’AFM souhaitent accélérer le processus d’enregistrement des thérapies dans les grandes agences de santé : l’ANSM en France, l’EMA à l’échelon européen et la FDA aux USA. L’objectif est de raccourcir les délais à deux ans avec l’introduction d’au moins une thérapie nouvelle par an. L’AFM et le Généthon ambitionnent aussi de faire en sorte que les traitements soient commercialisés à un coût raisonnable, d’où un travail sur la propriété intellectuelle et sur la relation avec les laboratoires pharmaceutiques qui industrialiseront la fabrication et la distribution des thérapies géniques. Une tâche ardue car il s’agira souvent de produits d’administration unique, comme une greffe, avec des modèles de revenus non récurrents. Ensuite, il faudra aussi transposer les méthodes et le savoir dans d’autres maladies rares comme la maladie de Huntington qui affecte le système nerveux et puis à terme celle de Parkinson. Et pourquoi pas, ultimement sur de nombreux cancers dont il semblerait qu’à terme ils puissent être traités plus efficacement par thérapies géniques.
Dans ma petite investigation sur cette série d’articles, j’ai été marqué par le côté très collaboratif et international de la recherche. Cela touche aussi bien au séquençage du génome humain (Human Genome Project) que le partage des données liées aux efforts qui ont suivi (1000genome, bases diverses mutualisées) que sur la recherche fondamentale sur le fonctionnement des cellules ou sur diverses thérapies. Les efforts du Généthon s’inscrivent dans ce cadre et ont eu et auront un impact majeur sur les progrès en matière de santé, très certainement largement au-delà des maladies rares. Les thérapies géniques sur les cancers pourraient en être un aboutissement mais il faudra encore de longues années avant d’y arriver.
Et le séquençage du génome dans tout ça ? C’était une étape lourde et utile qui a occupé le Généthon pendant les années 80 et 90. Mais une étape intermédiaire seulement. Ce qui a suivi s’est s’appuyé sur d’autres méthodes de recherche qui concernent le fonctionnement des cellules et la mécanique complexe des thérapies géniques. Le savoir faire cumulé dans le séquençage a cependant bien servi dans la création de thérapies géniques car les bases sont voisines : comment fonctionne la réplication de l’ADN, sa transposition en ARNm puis en protéines, comment l’ADN se recombine, se modifie, quels sont les éléments extérieurs (protéines, etc) qui conditionnent l’expression des gènes, etc. Dans le même temps, les avancées dans le savoir faire sur le séquençage a fait avancer l’outillage de laboratoire. Il y en a eu en parallèle sur les outils dits “biopuces” (DNA Arrays) qui ne font pas du séquençage mais de l’identification en masse de gènes ou polymorphismes dans l’ADN. Dans les deux cas, cet outillage est très utile pour accélérer la création de théraphies géniques, un long processus comme nous l’avons vu.
Pour nous réparer, nous qui ne sommes que poussière d’étoiles recyclée et assemblage biochimique… !
Making of
Ouf, fin des devoirs de vacances… ! Mais quel parcours passionnant.
Vous vous demandez peut-être comment j’ai pu m’y prendre pour rédiger tout ça sachant que je n’avais aucune connaissance préalable du sujet. Un petit “making of” s’impose en guise de chute.
Ici, pas de salon de visité ni de conférence. Le salon, c’est Internet ! Celui qui permet d’apprendre. Le point de départ de cette quête sont les articles de Wikipedia, de préférence en anglais, qui sont assez bien documentés et constituent une véritable fractale informationnelle. Puis Google Search pour affiner les recherches une fois identifiés les termes techniques clés du domaine.
La consultation des sites des fabricants était évidemment indispensable. Leur tendance à simplifier les choses pour des raisons marketing est compensée par les publications scientifiques qui accompagnent généralement toute introduction de nouveau procédé technique. Il existe de nombreux sites de publications scientifiques et heureusement, une part significative des articles y sont diffusés gratuitement. Il faut généralement s’accrocher pour les piger, d’où l’ingurgitation préalable d’un vocabulaire assez dense de biologie moléculaire. On trouve aussi parfois des thèses de doctorat très intéressantes qui brossent un tableau complet du sujet, le plus souvent en anglais.
Il existe aussi quelques supports de cours d’universités de médecine qui m’on servi de guides. A ceci près qu’ils ne sont pas toujours bien à la page. Ils traitent des principales méthodes de séquençage mais très peu des méthodes les plus récentes dites de troisième génération. C’est un peu le lot commun de l’enseignement supérieur lorsque la technologie évolue très vite. Côté illustrations, j’ai puisé également dans ces différentes sources mais je n’ai pas hésité à créer les miennes lorsque cela me semblait utile pour mieux vulgariser certains principes.
Mes articles sont aussi des fractales. Au départ, je me dis que je vais écrire “un article”. Et je me rend compte qu’il est trop grand. Je le coupe en deux. Quand j’avance dans la seconde partie, même constat et redécoupage en deux. Ainsi de suite jusqu’à épuiser le sujet, et ici, en sept parties. Pour cette dernière partie, mais en fait au début de ma série, j’ai fait une petite visite au Généthon et rencontré son Directeur Général, Frédéric Revah, qui m’a mis sur plusieurs pistes d’investigation et fourni les éléments d’informations relatif à cet épisode de la série. Puis quelques suggestions de lecteurs, eux-mêmes parfois entrepreneurs dans le domaine comme Patrick Merel de Portable Genomics.
Je voulais lever le voile sur ce qui était pour moi un véritable mystère technologique. Le voile est levé en grande partie. Mais ce n’est pas comme lorsque l’on découvre comment un tour de magie est réalisé, où l’on se dit “ah, c’était juste ça…” et que l’on ressort un peu déçu. Non. Ici, c’est différent. L’exploration de l’infiniment petit dans le vivant est un puits sans fonds. Le sujet est très complexe. J’ai été fasciné de découvrir tout ce que l’on savait mais aussi tout ce que l’on ne savait pas. La biogénétique est porteuse de nombreuses promesses qui vont sans conteste faire progresser notre santé, notre qualité de vie voire notre longévité dans de bonne conditions. Cela vaut le coup de se pencher dessus et ça vous change de la TV numérique que j’aborde abondamment dans ce blog !
Il est temps maintenant de revenir à une activité normale. La rentrée va me voir faire un tour habituel aux Universités d’Eté du MEDEF et je vais surtout ensuite visiter l’IBC à Amsterdam, ce salon étant la Mecque de la télévision numérique. Puis en octobre, seconde visite au CEATEC à Tokyo. Entre temps, je vais poursuivre ma série d’articles sur les français de la télévision connectée commencée en avril 2012. En plus de mon activité de conseil dans la TV numérique qui est la partie immergée de l’iceberg.
![]()
![]()
![]()
Reçevez par email les alertes de parution de nouveaux articles :
![]()
![]()
![]()


































 Articles
Articles
Les technologies de séquençage du génome humain – 7 http://t.co/ckhtb7BV #entrepreneur
J’ai tout lu.
Merci beaucoup Olivier !
Je ne trouve aucun labo français dans la liste des contributeurs à l’article de Nature “An integrated map of genetic variation from 1,092 human genomes” nov 2012.
Si aucun labo français ne participe au projet 1000 génomes, quelle en est la raison ?